分类: 人工智能
Swin Transformer 代码详解
code:https://github.com/microsoft/Swin-Transformer
代码详解: https://zhuanlan.zhihu.com/p/367111046

预处理:
对于分类模型,输入图像尺寸为 224×224×3 ,即 H=W=224 。按照原文描述,模型先将图像分割成每块大小为 4×4 的patch,那么就会有 56×56 个patch,这就是初始resolution,也是后面每个stage会降采样的维度。后面每个stage都会降采样时长宽降到一半,特征数加倍。按照原文及原图描述,划分的每个patch具有 4×4×3=48 维特征。
- 实际在代码中,首先使用了PatchEmbed模块(这里的PatchEmbed包括上图中的Linear Embedding 和 patch partition层),定义如下:
class PatchEmbed(nn.Module):
def __init__(self, img_size=224, patch_size=4, in_chans=3, embed_dim=96, norm_layer=None): # embed_dim就是上图中的C超参数
super().__init__()
img_size = to_2tuple(img_size)
patch_size = to_2tuple(patch_size)
patches_resolution = [img_size[0] // patch_size[0], img_size[1] // patch_size[1]]
self.img_size = img_size
self.patch_size = patch_size
self.patches_resolution = patches_resolution
self.num_patches = patches_resolution[0] * patches_resolution[1]
self.in_chans = in_chans
self.embed_dim = embed_dim
self.proj = nn.Conv2d(in_chans, embed_dim, kernel_size=patch_size, stride=patch_size)
if norm_layer is not None:
self.norm = norm_layer(embed_dim)
else:
self.norm = None
def forward(self, x):
B, C, H, W = x.shape
# FIXME look at relaxing size constraints
assert H == self.img_size[0] and W == self.img_size[1], \
f"Input image size ({H}*{W}) doesn't match model ({self.img_size[0]}*{self.img_size[1]})."
x = self.proj(x).flatten(2).transpose(1, 2) # B Ph*Pw C
if self.norm is not None:
x = self.norm(x)
return x可以看到,实际操作使用了一个卷积层conv2d(3, 96, 4, 4),直接就做了划分patch和编码初始特征的工作,对于输入 x:B×3×224×224 ,经过一层conv2d和LayerNorm得到 x:B×562×96 。然后作为对比,可以选择性地加上每个patch的绝对位置编码,原文实验表示这种做法不好,因此不会采用(ape=false)。最后经过一层dropout,至此,预处理完成。另外,要注意的是,代码和上面流程图并不符,其实在stage 1之前,即预处理完成后,维度已经是 H/4×W/4×C ,stage 1之后已经是 H/8×W/8×2C ,不过在stage 4后不再降采样,得到的还是 H/32×W/32×8C 。
stage处理
我们先梳理整个stage的大体过程,把简单的部分先说了,再深入到复杂得的细节。每个stage,即代码中的BasicLayer,由若干个block组成,而block的数目由depth列表中的元素决定。每个block就是W-MSA(window-multihead self attention)或者SW-MSA(shift window multihead self attention),一般有偶数个block,两种SA交替出现,比如6个block,0,2,4是W-MSA,1,3,5是SW-MSA。在经历完一个stage后,会进行下采样,定义的下采样比较有意思。比如还是 56×56 个patch,四个为一组,分别取每组中的左上,右上、左下、右下堆叠一起,经过一个layernorm,linear层,实现维度下采样、特征加倍的效果。实际上它可以看成一种加权池化的过程。代码如下:
class PatchMerging(nn.Module):
def __init__(self, input_resolution, dim, norm_layer=nn.LayerNorm):
super().__init__()
self.input_resolution = input_resolution
self.dim = dim
self.reduction = nn.Linear(4 * dim, 2 * dim, bias=False)
self.norm = norm_layer(4 * dim)
def forward(self, x):
"""
x: B, H*W, C
"""
H, W = self.input_resolution
B, L, C = x.shape
assert L == H * W, "input feature has wrong size"
assert H % 2 == 0 and W % 2 == 0, f"x size ({H}*{W}) are not even."
x = x.view(B, H, W, C)
x0 = x[:, 0::2, 0::2, :] # B H/2 W/2 C
x1 = x[:, 1::2, 0::2, :] # B H/2 W/2 C
x2 = x[:, 0::2, 1::2, :] # B H/2 W/2 C
x3 = x[:, 1::2, 1::2, :] # B H/2 W/2 C
x = torch.cat([x0, x1, x2, x3], -1) # B H/2 W/2 4*C
x = x.view(B, -1, 4 * C) # B H/2*W/2 4*C
x = self.norm(x)
x = self.reduction(x)
return x在经历完4个stage后,得到的是 (H/32×W/32)×8C 的特征,将其转到 8C×(H/32×W/32) 后,接一个AdaptiveAvgPool1d(1),全局平均池化,得到 8C 特征,最后接一个分类器。


Block处理
SwinTransformerBlock的结构,由LayerNorm层、windowAttention层(Window MultiHead self -attention, W-MSA)、MLP层以及shiftWindowAttention层(SW-MSA)组成。

上面说到有两种block,block的代码如下:
class SwinTransformerBlock(nn.Module):
r""" Swin Transformer Block.
Args:
dim (int): Number of input channels.
input_resolution (tuple[int]): Input resulotion.
num_heads (int): Number of attention heads.
window_size (int): Window size.
shift_size (int): Shift size for SW-MSA.
mlp_ratio (float): Ratio of mlp hidden dim to embedding dim.
qkv_bias (bool, optional): If True, add a learnable bias to query, key, value. Default: True
qk_scale (float | None, optional): Override default qk scale of head_dim ** -0.5 if set.
drop (float, optional): Dropout rate. Default: 0.0
attn_drop (float, optional): Attention dropout rate. Default: 0.0
drop_path (float, optional): Stochastic depth rate. Default: 0.0
act_layer (nn.Module, optional): Activation layer. Default: nn.GELU
norm_layer (nn.Module, optional): Normalization layer. Default: nn.LayerNorm
"""
def __init__(self, dim, input_resolution, num_heads, window_size=7, shift_size=0,
mlp_ratio=4., qkv_bias=True, qk_scale=None, drop=0., attn_drop=0., drop_path=0.,
act_layer=nn.GELU, norm_layer=nn.LayerNorm):
super().__init__()
self.dim = dim
self.input_resolution = input_resolution
self.num_heads = num_heads
self.window_size = window_size
self.shift_size = shift_size
self.mlp_ratio = mlp_ratio
if min(self.input_resolution) <= self.window_size:
# if window size is larger than input resolution, we don't partition windows
self.shift_size = 0
self.window_size = min(self.input_resolution)
assert 0 <= self.shift_size < self.window_size, "shift_size must in 0-window_size"
# 左图中最下边的LN层layerNorm层
self.norm1 = norm_layer(dim)
# W_MSA层或者SW-MSA层,详细的介绍看WindowAttention部分的代码
self.attn = WindowAttention(
dim, window_size=to_2tuple(self.window_size), num_heads=num_heads,
qkv_bias=qkv_bias, qk_scale=qk_scale, attn_drop=attn_drop, proj_drop=drop)
self.drop_path = DropPath(drop_path) if drop_path > 0. else nn.Identity()
# 左图中间部分的LN层
self.norm2 = norm_layer(dim)
mlp_hidden_dim = int(dim * mlp_ratio)
# 左图最上边的MLP层
self.mlp = Mlp(in_features=dim, hidden_features=mlp_hidden_dim, act_layer=act_layer, drop=drop)
# 这里利用shift_size控制是否执行shift window操作
# 当shift_size为0时,不执行shift操作,对应W-MSA,也就是在每个stage中,W-MSA与SW-MSA交替出现
# 例如第一个stage中存在两个block,那么第一个shift_size=0就是W-MSA,第二个shift_size不为0
# 就是SW-MSA
if self.shift_size > 0:
# calculate attention mask for SW-MSA
H, W = self.input_resolution
img_mask = torch.zeros((1, H, W, 1)) # 1 H W 1
#slice() 函数实现切片对象,主要用在切片操作函数里的参数传递。class slice(start, stop[, step])
h_slices = (slice(0, -self.window_size),
slice(-self.window_size, -self.shift_size),
slice(-self.shift_size, None))
w_slices = (slice(0, -self.window_size),
slice(-self.window_size, -self.shift_size),
slice(-self.shift_size, None))
cnt = 0
for h in h_slices:
for w in w_slices:
img_mask[:, h, w, :] = cnt
cnt += 1
## 上述操作是为了给每个窗口给上索引
mask_windows = window_partition(img_mask, self.window_size) # nW, window_size, window_size, 1
mask_windows = mask_windows.view(-1, self.window_size * self.window_size)
attn_mask = mask_windows.unsqueeze(1) - mask_windows.unsqueeze(2)
attn_mask = attn_mask.masked_fill(attn_mask != 0, float(-100.0)).masked_fill(attn_mask == 0, float(0.0))
else:
attn_mask = None
self.register_buffer("attn_mask", attn_mask)
def forward(self, x):
H, W = self.input_resolution
B, L, C = x.shape
assert L == H * W, "input feature has wrong size"
shortcut = x
x = self.norm1(x)
x = x.view(B, H, W, C)
# cyclic shift
# 如果需要计算 SW-MSA就需要进行循环移位。
if self.shift_size > 0:
shifted_x = torch.roll(x, shifts=(-self.shift_size, -self.shift_size), dims=(1, 2))
else:
shifted_x = x
# partition windows
x_windows = window_partition(shifted_x, self.window_size) # nW*B, window_size, window_size, C
x_windows = x_windows.view(-1, self.window_size * self.window_size, C) # nW*B, window_size*window_size, C
# W-MSA/SW-MSA
attn_windows = self.attn(x_windows, mask=self.attn_mask) # nW*B, window_size*window_size, C
# merge windows
attn_windows = attn_windows.view(-1, self.window_size, self.window_size, C)
shifted_x = window_reverse(attn_windows, self.window_size, H, W) # B H' W' C
# reverse cyclic shift
if self.shift_size > 0:
#shifts (python:int 或 tuple of python:int) —— 张量元素移位的位数。如果该参数是一个元组(例如shifts=(x,y)),dims必须是一个相同大小的元组(例如dims=(a,b)),相当于在第a维度移x位,在b维度移y位
x = torch.roll(shifted_x, shifts=(self.shift_size, self.shift_size), dims=(1, 2))
else:
x = shifted_x
x = x.view(B, H * W, C)
# FFN
x = shortcut + self.drop_path(x)
x = x + self.drop_path(self.mlp(self.norm2(x)))
return x
def extra_repr(self) -> str:
return f"dim={self.dim}, input_resolution={self.input_resolution}, num_heads={self.num_heads}, " \
f"window_size={self.window_size}, shift_size={self.shift_size}, mlp_ratio={self.mlp_ratio}"
def flops(self):
flops = 0
H, W = self.input_resolution
# norm1
flops += self.dim * H * W
# W-MSA/SW-MSA
nW = H * W / self.window_size / self.window_size
flops += nW * self.attn.flops(self.window_size * self.window_size)
# mlp
flops += 2 * H * W * self.dim * self.dim * self.mlp_ratio
# norm2
flops += self.dim * H * W
return flopsW-MSA
W-MSA比较简单,只要其中shift_size设置为0就是W-MSA。下面跟着代码走一遍过程。
- 输入: x:B×562×96 , H,W=56
- 经过一层layerNorm
- 变形: x:B×56×56×96
- 直接赋值给
shifted_x - 调用
window_partition函数,输入shifted_x,window_size=7: - 注意窗口大小以patch为单位,比如7就是7个patch,如果56的分辨率就会有8个窗口。
- 这个函数对
shifted_x做一系列变形,最终变成 82B×7×7×96 - 返回赋值给
x_windows,再变形成 82B×72×96 ,这表示所有图片,每个图片的64个window,每个window内有49个patch。 - 调用
WindowAttention层,这里以它的num_head为3为例。输入参数为x_windows和self.attn_mask,对于W-MSA,attn_mask为None,可以不用管。
WindowAttention代码如下:
代码中使用7×7的windowsize,将feature map分割为不同的window,在每个window中计算自注意力。
Self-attention的计算公式(B为相对位置编码)

绝对位置编码是在进行self-attention计算之前为每一个token添加一个可学习的参数,相对位置编码如上式所示,是在进行self-attention计算时,在计算过程中添加一个可学习的相对位置参数。
假设window_size = 2*2即每个窗口有4个token (M=2) ,如图1所示,在计算self-attention时,每个token都要与所有的token计算QK值,如图6所示,当位置1的token计算self-attention时,要计算位置1与位置(1,2,3,4)的QK值,即以位置1的token为中心点,中心点位置坐标(0,0),其他位置计算与当前位置坐标的偏移量。




最后生成的是相对位置索引,relative_position_index.shape = (M2,M2) ,在网络中注册成为一个不可学习的变量,relative_position_index的作用就是根据最终的索引值找到对应的可学习的相对位置编码。relative_position_index的数值范围(0~8),即 (2M−1)∗(2M−1) ,所以相对位置编码(relative position bias table)可以由一个3*3的矩阵表示,如图7所示:这样就根据index对应位置的索引找到table对应位置的值作为相对位置编码。
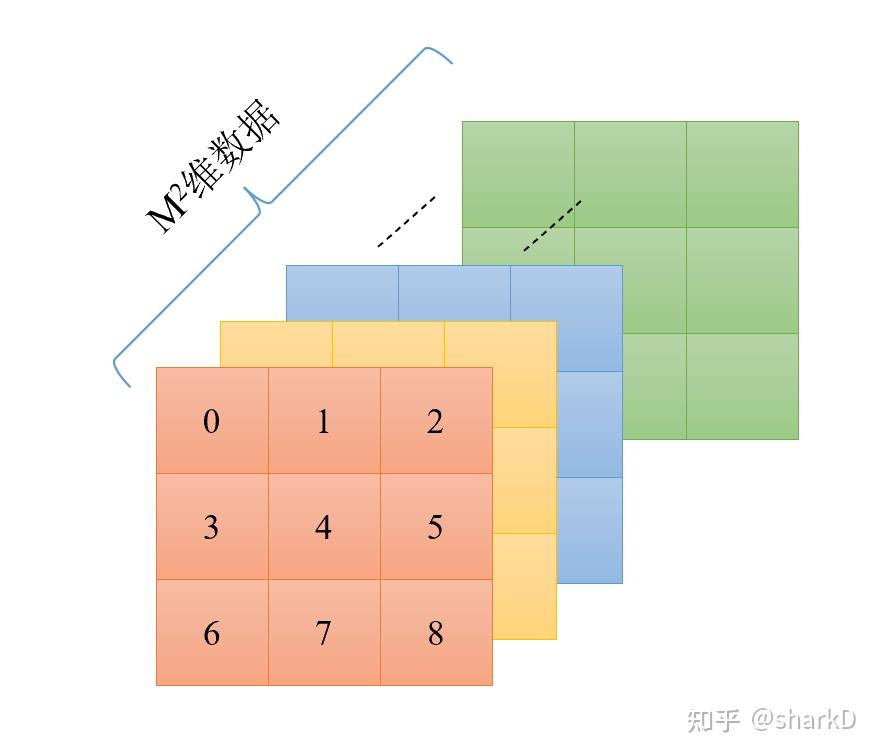

图7中的0-8为索引值,每个索引值都对应了 M2 维可学习数据(每个token都要计算 M2 个QK值,每个QK值都要加上对应的相对位置编码)
继续以图6中 M=2 的窗口为例,当计算位置1对应的 M2 个QK值时,应用的relative_position_index = [ 4, 5, 7, 8] (M2)个 ,对应的数据就是图7中位置索引4,5,7,8位置对应的 M2 维数据,即relative_position.shape = (M2∗M2)
相对位置编码在源码WindowAttention中应用,了解原理之后就很容易能够读懂程序:
class WindowAttention(nn.Module):
r""" Window based multi-head self attention (W-MSA) module with relative position bias.
It supports both of shifted and non-shifted window.
Args:
dim (int): Number of input channels.
window_size (tuple[int]): The height and width of the window.
num_heads (int): Number of attention heads.
qkv_bias (bool, optional): If True, add a learnable bias to query, key, value. Default: True
qk_scale (float | None, optional): Override default qk scale of head_dim ** -0.5 if set
attn_drop (float, optional): Dropout ratio of attention weight. Default: 0.0
proj_drop (float, optional): Dropout ratio of output. Default: 0.0
"""
def __init__(self, dim, window_size, num_heads, qkv_bias=True, qk_scale=None, attn_drop=0., proj_drop=0.):
super().__init__()
self.dim = dim # 输入通道的数量
self.window_size = window_size # Wh, Ww
self.num_heads = num_heads
head_dim = dim // num_heads
self.scale = qk_scale or head_dim ** -0.5
# define a parameter table of relative position bias
self.relative_position_bias_table = nn.Parameter(
torch.zeros((2 * window_size[0] - 1) * (2 * window_size[1] - 1), num_heads)) # 2*Wh-1 * 2*Ww-1, nH 初始化表
# get pair-wise relative position index for each token inside the window
coords_h = torch.arange(self.window_size[0]) # coords_h = tensor([0,1,2,...,self.window_size[0]-1]) 维度=Wh
coords_w = torch.arange(self.window_size[1]) # coords_w = tensor([0,1,2,...,self.window_size[1]-1]) 维度=Ww
coords = torch.stack(torch.meshgrid([coords_h, coords_w])) # 2, Wh, Ww
coords_flatten = torch.flatten(coords, 1) # 2, Wh*Ww
relative_coords = coords_flatten[:, :, None] - coords_flatten[:, None, :] # 2, Wh*Ww, Wh*Ww
relative_coords = relative_coords.permute(1, 2, 0).contiguous() # Wh*Ww, Wh*Ww, 2
relative_coords[:, :, 0] += self.window_size[0] - 1 # shift to start from 0
relative_coords[:, :, 1] += self.window_size[1] - 1
'''
后面我们需要将其展开成一维偏移量。而对于(2,1)和(1,2)这两个坐标,在二维上是不同的,但是通过将x\y坐标相加转换为一维偏移的时候
他们的偏移量是相等的,所以需要对其做乘法操作,进行区分
'''
relative_coords[:, :, 0] *= 2 * self.window_size[1] - 1
# 计算得到相对位置索引
# relative_position_index.shape = (M2, M2) 意思是一共有这么多个位置
relative_position_index = relative_coords.sum(-1) # Wh*Ww, Wh*Ww
'''
relative_position_index注册为一个不参与网络学习的变量
'''
self.register_buffer("relative_position_index", relative_position_index)
self.qkv = nn.Linear(dim, dim * 3, bias=qkv_bias)
self.attn_drop = nn.Dropout(attn_drop)
self.proj = nn.Linear(dim, dim)
self.proj_drop = nn.Dropout(proj_drop)
'''
使用从截断正态分布中提取的值填充输入张量
self.relative_position_bias_table 是全0张量,通过trunc_normal_ 进行数值填充
'''
trunc_normal_(self.relative_position_bias_table, std=.02)
self.softmax = nn.Softmax(dim=-1)
def forward(self, x, mask=None):
"""
Args:
x: input features with shape of (num_windows*B, N, C)
N: number of all patches in the window
C: 输入通过线性层转化得到的维度C
mask: (0/-inf) mask with shape of (num_windows, Wh*Ww, Wh*Ww) or None
"""
B_, N, C = x.shape
'''
x.shape = (num_windows*B, N, C)
self.qkv(x).shape = (num_windows*B, N, 3C)
self.qkv(x).reshape(B_, N, 3, self.num_heads, C // self.num_heads).shape = (num_windows*B, N, 3, num_heads, C//num_heads)
self.qkv(x).reshape(B_, N, 3, self.num_heads, C // self.num_heads).permute(2, 0, 3, 1, 4).shape = (3, num_windows*B, num_heads, N, C//num_heads)
'''
qkv = self.qkv(x).reshape(B_, N, 3, self.num_heads, C // self.num_heads).permute(2, 0, 3, 1, 4)
'''
q.shape = k.shape = v.shape = (num_windows*B, num_heads, N, C//num_heads)
N = M2 代表patches的数量
C//num_heads代表Q,K,V的维数
'''
q, k, v = qkv[0], qkv[1], qkv[2] # make torchscript happy (cannot use tensor as tuple)
# q乘上一个放缩系数,对应公式中的sqrt(d)
q = q * self.scale
# attn.shape = (num_windows*B, num_heads, N, N) N = M2 代表patches的数量
attn = (q @ k.transpose(-2, -1))
'''
self.relative_position_bias_table.shape = (2*Wh-1 * 2*Ww-1, nH)
self.relative_position_index.shape = (Wh*Ww, Wh*Ww)
self.relative_position_index矩阵中的所有值都是从self.relative_position_bias_table中取的
self.relative_position_index是计算出来不可学习的量
'''
relative_position_bias = self.relative_position_bias_table[self.relative_position_index.view(-1)].view(
self.window_size[0] * self.window_size[1], self.window_size[0] * self.window_size[1], -1) # Wh*Ww,Wh*Ww,nH
relative_position_bias = relative_position_bias.permute(2, 0, 1).contiguous() # nH, Wh*Ww, Wh*Ww
'''
attn.shape = (num_windows*B, num_heads, M2, M2) N = M2 代表patches的数量
.unsqueeze(0):扩张维度,在0对应的位置插入维度1
relative_position_bias.unsqueeze(0).shape = (1, num_heads, M2, M2)
num_windows*B 通过广播机制传播,relative_position_bias.unsqueeze(0).shape = (1, nH, M2, M2) 的维度1会broadcast到数量num_windows*B
表示所有batch通用一个索引矩阵和相对位置矩阵
'''
attn = attn + relative_position_bias.unsqueeze(0)
# mask.shape = (num_windows, M2, M2)
# attn.shape = (num_windows*B, num_heads, M2, M2)
if mask is not None:
nW = mask.shape[0]
# attn.view(B_ // nW, nW, self.num_heads, N, N).shape = (B, num_windows, num_heads, M2, M2) 第一个M2代表有M2个token,第二个M2代表每个token要计算M2次QKT的值
# mask.unsqueeze(1).unsqueeze(0).shape = (1, num_windows, 1, M2, M2) 第一个M2代表有M2个token,第二个M2代表每个token要计算M2次QKT的值
# broadcast相加
attn = attn.view(B_ // nW, nW, self.num_heads, N, N) + mask.unsqueeze(1).unsqueeze(0)
# attn.shape = (B, num_windows, num_heads, M2, M2)
attn = attn.view(-1, self.num_heads, N, N)
attn = self.softmax(attn)
else:
attn = self.softmax(attn)
attn = self.attn_drop(attn)
'''
v.shape = (num_windows*B, num_heads, M2, C//num_heads) N=M2 代表patches的数量, C//num_heads代表输入的维度
attn.shape = (num_windows*B, num_heads, M2, M2)
attn@v .shape = (num_windows*B, num_heads, M2, C//num_heads)
'''
x = (attn @ v).transpose(1, 2).reshape(B_, N, C) # B_:num_windows*B N:M2 C=num_heads*C//num_heads
# self.proj = nn.Linear(dim, dim) dim = C
# self.proj_drop = nn.Dropout(proj_drop)
x = self.proj(x)
x = self.proj_drop(x)
return x # x.shape = (num_windows*B, N, C) N:窗口中所有patches的数量
def extra_repr(self) -> str:
return f'dim={self.dim}, window_size={self.window_size}, num_heads={self.num_heads}'
def flops(self, N):
# calculate flops for 1 window with token length of N
flops = 0
# qkv = self.qkv(x)
flops += N * self.dim * 3 * self.dim
# attn = (q @ k.transpose(-2, -1))
flops += self.num_heads * N * (self.dim // self.num_heads) * N
# x = (attn @ v)
flops += self.num_heads * N * N * (self.dim // self.num_heads)
# x = self.proj(x)
flops += N * self.dim * self.dim
return flops在上述程序中有一段mask相关程序:
if mask is not None:
nW = mask.shape[0]
# attn.view(B_ // nW, nW, self.num_heads, N, N).shape = (B, num_windows, num_heads, M2, M2) 第一个M2代表有M2个token,第二个M2代表每个token要计算M2次QKT的值
# mask.unsqueeze(1).unsqueeze(0).shape = (1, num_windows, 1, M2, M2) 第一个M2代表有M2个token,第二个M2代表每个token要计算M2次QKT的值
# broadcast相加
attn = attn.view(B_ // nW, nW, self.num_heads, N, N) + mask.unsqueeze(1).unsqueeze(0)
# attn.shape = (B, num_windows, num_heads, M2, M2)
attn = attn.view(-1, self.num_heads, N, N)
attn = self.softmax(attn)
else:
attn = self.softmax(attn)这个部分对应的是Swin Transformer Block 中的SW-MSA
- 输入 x:82B×72×96 。
- 产生 QKV ,调用线性层后,得到 82B×72×(96×3) ,拆分给不同的head,得到 82B×72×3×3×32 ,第一个3是 QKV 的3,第二个3是3个head。再permute成 3×82B×3×72×32 ,再拆解成 q,k,v ,每个都是 82B×3×72×32 。表示所有图片的每个图片64个window,每个window对应到3个不同的head,都有一套49个patch、32维的特征。
- q 归一化
- qk 矩阵相乘求特征内积,得到 attn:82B×3×72×72
- 得到相对位置的编码信息
relative_position_bias:- 代码如下:
self.relative_position_bias_table = nn.Parameter(
torch.zeros((2 * window_size[0] - 1) * (2 * window_size[1] - 1), num_heads)) # 2*Wh-1 * 2*Ww-1, nH
# get pair-wise relative position index for each token inside the window
coords_h = torch.arange(self.window_size[0])
coords_w = torch.arange(self.window_size[1])
coords = torch.stack(torch.meshgrid([coords_h, coords_w])) # 2, Wh, Ww
coords_flatten = torch.flatten(coords, 1) # 2, Wh*Ww
relative_coords = coords_flatten[:, :, None] - coords_flatten[:, None, :] # 2, Wh*Ww, Wh*Ww
relative_coords = relative_coords.permute(1, 2, 0).contiguous() # Wh*Ww, Wh*Ww, 2
relative_coords[:, :, 0] += self.window_size[0] - 1 # shift to start from 0
relative_coords[:, :, 1] += self.window_size[1] - 1
relative_coords[:, :, 0] *= 2 * self.window_size[1] - 1
relative_position_index = relative_coords.sum(-1) # Wh*Ww, Wh*Ww
self.register_buffer("relative_position_index", relative_position_index)- 这里以
window_size=3为例,解释以下过程:首先生成 coords:2×3×3 ,就是在一个 3×3 的窗口内,每个位置的 y,x 坐标,而relative_coords为 2×9×9 ,就是9个点中,每个点的 y 或 x 与其他所有点的差值,比如 [0][3][1] 表示3号点(第二行第一个点)与1号点(第一行第二个点)的 y 坐标的差值。然后变形,并让两个坐标分别加上 3−1=2 ,是因为这些坐标值范围 [0,2] ,因此差值的最小值为-2,加上2后从0开始。最后让 y 坐标乘上 2×3−1=5 ,应该是一个trick,调整差值范围。最后将两个维度的差值相加,得到relative_position_index, 32×32 ,为9个点之间两两之间的相对位置编码值,最后用来到self.relative_position_bias_table中寻址,注意相对位置的最大值为 (2M−2)(2M−1) ,而这个table最多有 (2M−1)(2M−1) 行,因此保证可以寻址,得到了一组给多个head使用的相对位置编码信息,这个table是可训练的参数。 - 回到代码中,得到的
relative_position_bias为 3×72×72 - 将其加到
attn上,最后一个维度softmax,dropout - 与 v 矩阵相乘,并转置,合并多个头的信息,得到 82B×72×96
- 经过一层线性层,dropout,返回
- 返回赋值给
attn_windows,变形为 82B×7×7×96 - 调用
window_reverse,打回原状: B×56×56×96 - 返回给 x ,经过FFN:先加上原来的输入 x 作为residue结构,注意这里用到timm的
DropPath,并且drop的概率是整个网络结构线性增长的。然后再加上两层mlp的结果。 - 返回结果 x 。
这样,整个过程就完成了,剩下的就是SW-MSA的一些不同的操作。

- 首先将windows进行半个窗口的循环移位,上图中的1, 2步骤,使用torch.roll实现。
- 在相同的窗口中计算自注意力,计算结果如下右图所示,window0的结构保存,但是针对window2的计算,其中3与3、6与6的计算生成了attn mask 中window2中的黄色区域,针对windows2中3与6、6与3之间不应该计算自注意力(attn mask中window2的蓝色区域),将蓝色区域mask赋值为-100,经过softmax之后,起作用可以忽略不计。同理window1与window3的计算一致。
- 最后再进行循环移位,恢复原来的位置。

原论文图中的Stage和程序中的一个Stage不同:
程序中的BasicLayer为一个Stage,在BasicLayer中调用了上面讲到的SwinTransformerBlock和PatchMerging模块:
class BasicLayer(nn.Module): # 论文图中每个stage里对应的若干个SwinTransformerBlock
""" A basic Swin Transformer layer for one stage.
Args:
dim (int): Number of input channels.
input_resolution (tuple[int]): Input resolution.
depth (int): Number of blocks.
num_heads (int): Number of attention heads.
window_size (int): Local window size.
mlp_ratio (float): Ratio of mlp hidden dim to embedding dim.
qkv_bias (bool, optional): If True, add a learnable bias to query, key, value. Default: True
qk_scale (float | None, optional): Override default qk scale of head_dim ** -0.5 if set.
drop (float, optional): Dropout rate. Default: 0.0
attn_drop (float, optional): Attention dropout rate. Default: 0.0
drop_path (float | tuple[float], optional): Stochastic depth rate. Default: 0.0
norm_layer (nn.Module, optional): Normalization layer. Default: nn.LayerNorm
downsample (nn.Module | None, optional): Downsample layer at the end of the layer. Default: None
use_checkpoint (bool): Whether to use checkpointing to save memory. Default: False.
"""
def __init__(self, dim, input_resolution, depth, num_heads, window_size,
mlp_ratio=4., qkv_bias=True, qk_scale=None, drop=0., attn_drop=0.,
drop_path=0., norm_layer=nn.LayerNorm, downsample=None, use_checkpoint=False):
super().__init__()
self.dim = dim
self.input_resolution = input_resolution
self.depth = depth # swin_transformer blocks的个数
self.use_checkpoint = use_checkpoint
# build blocks 从0开始的偶数位置的SwinTransformerBlock计算的是W-MSA,奇数位置的Block计算的是SW-MSA,且shift_size = window_size//2
self.blocks = nn.ModuleList([
SwinTransformerBlock(dim=dim, input_resolution=input_resolution,
num_heads=num_heads, window_size=window_size,
shift_size=0 if (i % 2 == 0) else window_size // 2,
mlp_ratio=mlp_ratio,
qkv_bias=qkv_bias, qk_scale=qk_scale,
drop=drop, attn_drop=attn_drop,
drop_path=drop_path[i] if isinstance(drop_path, list) else drop_path,
norm_layer=norm_layer)
for i in range(depth)])
# patch merging layer
if downsample is not None:
self.downsample = downsample(input_resolution, dim=dim, norm_layer=norm_layer)
else:
self.downsample = None
def forward(self, x):
for blk in self.blocks:
if self.use_checkpoint:
x = checkpoint.checkpoint(blk, x)
else:
x = blk(x) # blk = SwinTransformerBlock
if self.downsample is not None:
x = self.downsample(x)
return x
def extra_repr(self) -> str:
return f"dim={self.dim}, input_resolution={self.input_resolution}, depth={self.depth}"
def flops(self):
flops = 0
for blk in self.blocks:
flops += blk.flops()
if self.downsample is not None:
flops += self.downsample.flops()
return flopsPart 3 : 不同视觉任务输出
程序中对应的是图片分类任务,经过Part 2 之后的数据通过 norm/avgpool/flatten:
x = self.norm(x) # B L C
x = self.avgpool(x.transpose(1, 2)) # B C 1
x = torch.flatten(x, 1) # B C之后通过nn.Linear将特征转化为对应的类别:
self.head = nn.Linear(self.num_features, num_classes) if num_classes > 0 else nn.Identity()应用于其他不同的视觉任务时,只需要将输出进行特定的修改即可。
完整的SwinTransformer程序如下:
class SwinTransformer(nn.Module):
r""" Swin Transformer
A PyTorch impl of : `Swin Transformer: Hierarchical Vision Transformer using Shifted Windows` -
https://arxiv.org/pdf/2103.14030
Args:
img_size (int | tuple(int)): Input image size. Default 224
patch_size (int | tuple(int)): Patch size. Default: 4
in_chans (int): Number of input image channels. Default: 3
num_classes (int): Number of classes for classification head. Default: 1000
embed_dim (int): Patch embedding dimension. Default: 96
depths (tuple(int)): Depth of each Swin Transformer layer.
num_heads (tuple(int)): Number of attention heads in different layers.
window_size (int): Window size. Default: 7
mlp_ratio (float): Ratio of mlp hidden dim to embedding dim. Default: 4
qkv_bias (bool): If True, add a learnable bias to query, key, value. Default: True
qk_scale (float): Override default qk scale of head_dim ** -0.5 if set. Default: None
drop_rate (float): Dropout rate. Default: 0
attn_drop_rate (float): Attention dropout rate. Default: 0
drop_path_rate (float): Stochastic depth rate. Default: 0.1
norm_layer (nn.Module): Normalization layer. Default: nn.LayerNorm.
ape (bool): If True, add absolute position embedding to the patch embedding. Default: False
patch_norm (bool): If True, add normalization after patch embedding. Default: True
use_checkpoint (bool): Whether to use checkpointing to save memory. Default: False
"""
def __init__(self, img_size=224, patch_size=4, in_chans=3, num_classes=1000,
embed_dim=96, depths=[2, 2, 6, 2], num_heads=[3, 6, 12, 24],
window_size=7, mlp_ratio=4., qkv_bias=True, qk_scale=None,
drop_rate=0., attn_drop_rate=0., drop_path_rate=0.1,
norm_layer=nn.LayerNorm, ape=False, patch_norm=True,
use_checkpoint=False, **kwargs):
super().__init__()
self.num_classes = num_classes # 1000
self.num_layers = len(depths) # [2, 2, 6, 2] Swin_T 的配置
self.embed_dim = embed_dim # 96
self.ape = ape # False
self.patch_norm = patch_norm # True
self.num_features = int(embed_dim * 2 ** (self.num_layers - 1)) # 96*2^3
self.mlp_ratio = mlp_ratio # 4
# split image into non-overlapping patches
self.patch_embed = PatchEmbed(
img_size=img_size, patch_size=patch_size, in_chans=in_chans, embed_dim=embed_dim,
norm_layer=norm_layer if self.patch_norm else None)
num_patches = self.patch_embed.num_patches
patches_resolution = self.patch_embed.patches_resolution
self.patches_resolution = patches_resolution
# absolute position embedding
if self.ape:
self.absolute_pos_embed = nn.Parameter(torch.zeros(1, num_patches, embed_dim))
trunc_normal_(self.absolute_pos_embed, std=.02)
self.pos_drop = nn.Dropout(p=drop_rate)
# stochastic depth
dpr = [x.item() for x in torch.linspace(0, drop_path_rate, sum(depths))] # stochastic depth decay rule
# build layers
self.layers = nn.ModuleList()
for i_layer in range(self.num_layers):
layer = BasicLayer(dim=int(embed_dim * 2 ** i_layer),
input_resolution=(patches_resolution[0] // (2 ** i_layer),
patches_resolution[1] // (2 ** i_layer)),
depth=depths[i_layer],
num_heads=num_heads[i_layer],
window_size=window_size,
mlp_ratio=self.mlp_ratio,
qkv_bias=qkv_bias, qk_scale=qk_scale,
drop=drop_rate, attn_drop=attn_drop_rate,
drop_path=dpr[sum(depths[:i_layer]):sum(depths[:i_layer + 1])],
norm_layer=norm_layer,
downsample=PatchMerging if (i_layer < self.num_layers - 1) else None,
use_checkpoint=use_checkpoint)
self.layers.append(layer)
self.norm = norm_layer(self.num_features) # norm_layer = nn.LayerNorm
self.avgpool = nn.AdaptiveAvgPool1d(1)
self.head = nn.Linear(self.num_features, num_classes) if num_classes > 0 else nn.Identity()
self.apply(self._init_weights) # 使用self.apply 初始化参数
def _init_weights(self, m):
# is_instance 判断对象是否为已知类型
if isinstance(m, nn.Linear):
trunc_normal_(m.weight, std=.02)
if isinstance(m, nn.Linear) and m.bias is not None:
nn.init.constant_(m.bias, 0)
elif isinstance(m, nn.LayerNorm):
nn.init.constant_(m.bias, 0)
nn.init.constant_(m.weight, 1.0)
@torch.jit.ignore
def no_weight_decay(self):
return {'absolute_pos_embed'}
@torch.jit.ignore
def no_weight_decay_keywords(self):
return {'relative_position_bias_table'}
def forward_features(self, x):
x = self.patch_embed(x) # x.shape = (H//4, W//4, C)
if self.ape:
x = x + self.absolute_pos_embed
x = self.pos_drop(x) # self.pos_drop = nn.Dropout(p=drop_rate)
for layer in self.layers:
x = layer(x)
x = self.norm(x) # B L C
x = self.avgpool(x.transpose(1, 2)) # B C 1
x = torch.flatten(x, 1) # B C
return x
def forward(self, x):
x = self.forward_features(x) # x是论文图中Figure 3 a图中最后的输出
# self.head = nn.Linear(self.num_features, num_classes) if num_classes > 0 else nn.Identity()
x = self.head(x) # x.shape = (B, num_classes)
return x
def flops(self):
flops = 0
flops += self.patch_embed.flops()
for i, layer in enumerate(self.layers):
flops += layer.flops()
flops += self.num_features * self.patches_resolution[0] * self.patches_resolution[1] // (2 ** self.num_layers)
flops += self.num_features * self.num_classes
return flops补充:有关swin transformer相对位置编码:




VIT

Dosovitskiy et al. An image is worth 16×16 words: transformers for image recognition at scale. In ICLR, 2021
step1 :分割图片

step2 向量化:从九个快变成九个向量

step3:向量线性变换:(linear embedding线性嵌入层)

step4:将位置编码添加到z上:

step4:添加一个cls向量:

step5:只利用cls的输出

按照上面的流程图,一个ViT block可以分为以下几个步骤
(1) patch embedding:例如输入图片大小为224×224,将图片分为固定大小的patch,patch大小为16×16,则每张图像会生成224×224/16×16=196个patch,即输入序列长度为196,每个patch维度16x16x3=768,线性投射层的维度为768xN (N=768),因此输入通过线性投射层之后的维度依然为196×768,即一共有196个token,每个token的维度是768。这里还需要加上一个特殊字符cls,因此最终的维度是197×768。到目前为止,已经通过patch embedding将一个视觉问题转化为了一个seq2seq问题
(2) positional encoding(standard learnable 1D position embeddings):ViT同样需要加入位置编码,位置编码可以理解为一张表,表一共有N行,N的大小和输入序列长度相同,每一行代表一个向量,向量的维度和输入序列embedding的维度相同(768)。注意位置编码的操作是sum,而不是concat。加入位置编码信息之后,维度依然是197×768
(3) LN/multi-head attention/LN:LN输出维度依然是197×768。多头自注意力时,先将输入映射到q,k,v,如果只有一个头,qkv的维度都是197×768,如果有12个头(768/12=64),则qkv的维度是197×64,一共有12组qkv,最后再将12组qkv的输出拼接起来,输出维度是197×768,然后在过一层LN,维度依然是197×768
(4) MLP:将维度放大再缩小回去,197×768放大为197×3072,再缩小变为197×768
一个block之后维度依然和输入相同,都是197×768,因此可以堆叠多个block。最后会将特殊字符cls对应的输出 zL0 作为encoder的最终输出 ,代表最终的image presentation(另一种做法是不加cls字符,对所有的tokens的输出做一个平均),如下图公式(4),后面接一个MLP进行图片分类
vit需要预训练+微调


• Pretrain the model on Dataset A, fine-tune the model on Dataset B,
and evaluate the model on Dataset B.
• Pretrained on ImageNet (small), ViT is slightly worse than ResNet.
• Pretrained on ImageNet-21K (medium), ViT is comparable to ResNet.
• Pretrained on JFT (large), ViT is slightly better than ResNet.
效果:

Swin Transformer论文解读与思考
论文: https://arxiv.org/abs/2103.14030
github:https://github.com/microsoft/Swin-Transformer
代码详解 https://zhuanlan.zhihu.com/p/384514268
Vision Transformer , Vision MLP 超详细解读 (原理分析+代码解读) (目录)
论文详解:https://space.bilibili.com/1567748478/channel/collectiondetail?sid=32744
Swin Transformer视频讲解:
https://github.com/WZMIAOMIAO/deep-learning-for-image-processing
摘要 :目前transformer应用于CV领域的挑战主要有两个,一个是图片多尺度语义信息的问题,同一个物体在不同图片中的大小尺度变化很大,另外就是难以处理高分辨的图片,如果以pix像素作为序列元素,那么计算成本太大,因此一部分方法是将CNN提取图片特征在送进transformer中 ,或者通过patch,将图片变成一个个的patch。作者提出Swin Transformer 目标是希望作为一种计算机视觉的通用主干网络(因为VIT的提出已经证明了Transformer在CV的可行性),这是一种层级的架构。通过窗口注意力以及转移窗口注意力,不仅降低了计算量,同时层级架构对于不同尺度的信息处理都十分灵活,该架构在图像分类、目标检测、语义分割等任务中表现出色。(对于图像分类、目标检测、语义分割等下游任务,尤其是密集预测任务,多尺度特征是十分必要的)
引言
首先来看作者给出的Swin Transformer 和 VIT结构对比:

VIT的patch固定16*16(可以认为是16倍下采样),多尺度特征处理不好,因为整个过程都是在同一尺度下操作的,出来的特征是单尺度的,优点是全局的特征处理比较强,因为是在全局的尺度进行操作的,但因此他的复杂度跟图像尺寸成平方倍的增长,很难处理目前图像分割检测。再来看 Swin Transformer ,通过图可以看出作者借鉴了CNN的很多设计思路,为了减少序列长度,减低计算量,仅在上面的红框中进行自注意力计算,计算复杂度会跟整张图片的大小成线性关系。另外作者使用基于窗口的注意力的也可以很好的把握物体的全局信息(因为在CV中,一个物体的绝大部分都存在单个windows窗口中,很少会横跨多个窗口),另外CNN网络的如何抓住物体的多尺度特征?是因为pool池化层的存在,每次池化能够增大卷积核看到的感受野。因此作者提出了patch merging,将相邻的四个patch合并成一个大patch(可以认为是加权池化),这样合并出来的一个 大patch就可以看到四个小patch内容,感受野增大。有了多尺度特征(4*,8*,16*多尺度特征图)以后,可以接一个FPN头,由于做检测任务,也可以放在unet做分割任务,这就是作者所说的, Swin Transformer 是可以做一个通用骨干网络。
Transformer的初衷就是更好的理解上下文,如果窗口都是不重叠的,那自注意力真的就变成孤立自注意力,就没有全局建模的能力
Swin Transformer 的一个关键设计因素:移动窗口操作。在第l层,通过划分不同的小窗口(实际中是一个窗口有7*7个patch(最小单位),这里示意图以4*4的patch作为一个窗口),自注意力只在窗口中计算 ,就可以有效降低序列长度,从而减少计算复杂度。shift操作可以认为是将l层的窗口整体向右下加移动两个patch所形成的新的窗口,新的特征图进行分割windws以后就有l+1层所示的这些窗口(如下图共九个)了。如果没有shift,那么所有窗口不重叠,在窗口进行自注意力时候,窗口之间无法交互,就无法达到transformer的初衷了(更好的理解上下文),shift后不同窗口的patch就可以进行交互了。再加上一个patch merging操作,不断扩大感受野,到最后几层的时候,每个patch的感受野已经很大了,实际上就可以看到大部分图片了,shift操作以后,就可以看成是全局注意力操作,这样即省内存效果也好。

引言的最后,作者坚信,一个CV和NLP大一统的框架是可以促进两个领域共同发展的,但实际上 Swin Transformer 更多的是利用了CNN的先验知识,从而在计算机视觉领域大杀四方。但是在模型大一统上,也就是 unified architecture 上来说,其实 ViT 还是做的更好的,因为它真的可以什么都不改,什么先验信息都不加,就能让Transformer在两个领域都能用的很好,这样模型不仅可以共享参数,而且甚至可以把所有模态的输入直接就拼接起来,当成一个很长的输入,直接扔给Transformer去做,而不用考虑每个模态的特性
先看结论:
这篇论文提出了 Swin Transformer,它是一个层级式的Transformer,而且它的计算复杂度是跟输入图像的大小呈线性增长的。Swin Transformerr 在 COCO 和 ADE20K上的效果都非常的好,远远超越了之前最好的方法,所以作者说基于此,希望 Swin Transformer 能够激发出更多更好的工作,尤其是在多模态方面。
因为在Swin Transformer 这篇论文里最关键的一个贡献就是基于 Shifted Window 的自注意力,它对很多视觉的任务,尤其是对下游密集预测型的任务是非常有帮助的,但是如果 Shifted Window 操作不能用到 NLP 领域里,其实在模型大一统上论据就不是那么强了,所以作者说接下来他们的未来工作就是要把 Shifted Windows用到 NLP 里面,而且如果真的能做到这一点,那 Swin Transformer真的就是一个里程碑式的工作了,而且模型大一统的故事也就讲的圆满了
方法
主要分为两大块
- 大概把整体的流程讲了一下,主要就是过了一下前向过程,以及提出的 patch merging 操作是怎么做的
- 基于 Shifted Window 的自注意力,Swin Transformer怎么把它变成一个transformer block 进行计算
前向过程
- 假设说有一张224*224*3(ImageNet 标准尺寸)的输入图片
- 第一步就是像 ViT 那样把图片打成 patch,在 Swin Transformer 这篇论文里,它的 patch size 是4*4,而不是像 ViT 一样16*16,所以说它经过 patch partition 打成 patch 之后,得到图片的尺寸是56*56*48,56就是224/4,因为 patch size 是4,向量的维度48,因为4*4*3,3 是图片的 RGB 通道
- 打完了 patch ,接下来就要做 Linear Embedding,也就是说要把向量的维度变成一个预先设置好的值,就是 Transformer 能够接受的值,在 Swin Transformer 的论文里把这个超参数设为 c,对于 Swin tiny 网络来说,也就是上图中画的网络总览图,它的 c 是96,所以经历完 Linear Embedding 之后,输入的尺寸就变成了56*56*96,前面的56*56就会拉直变成3136,变成了序列长度,后面的96就变成了每一个token向量的维度,其实 Patch Partition 和 Linear Embedding 就相当于是 ViT 里的Patch Projection 操作,而在代码里也是用一次卷积操作就完成了,
- 第一部分跟 ViT 其实还是没有区别的,但紧接着区别就来了
- 首先序列长度是3136,对于 ViT 来说,用 patch size 16*16,它的序列长度就只有196,是相对短很多的,这里的3136就太长了,是目前来说Transformer不能接受的序列长度,所以 Swin Transformer 就引入了基于窗口的自注意力计算,每个窗口按照默认来说,都只有七七四十九个 patch,所以说序列长度就只有49就相当小了,这样就解决了计算复杂度的问题
- 所以也就是说, stage1中的swin transformer block 是基于窗口计算自注意力的,现在暂时先把 transformer block当成是一个黑盒,只关注输入和输出的维度,对于 Transformer 来说,如果不对它做更多约束的话,Transformer输入的序列长度是多少,输出的序列长度也是多少,它的输入输出的尺寸是不变的,所以说在 stage1 中经过两层Swin Transformer block 之后,输出还是56*56*96
- 到这其实 Swin Transformer的第一个阶段就走完了,也就是先过一个 Patch Projection 层,然后再过一些 Swin Transformer block,接下来如果想要有多尺寸的特征信息,就要构建一个层级式的 transformer,也就是说需要一个像卷积神经网络里一样,有一个类似于池化的操作

Patch Merging
Patch Merging 其实在之前一些工作里也有用到,它很像 Pixel Shuffle 的上采样的一个反过程,Pixel Shuffle 是 lower level 任务中很常用的一个上采样方式
- 假如有一个张量, Patch Merging 顾名思义就是把临近的小 patch 合并成一个大 patch,这样就可以起到下采样一个特征图的效果了
- 这里因为是想下采样两倍,所以说在选点的时候是每隔一个点选一个,也就意味着说对于这个张量来说,每次选的点是1、1、1、1
- 其实在这里的1、2、3、4并不是矩阵里有的值,而是给它的一个序号,同样序号位置上的 patch 就会被 merge 到一起,这个序号只是为了帮助理解
- 经过隔一个点采一个样之后,原来的这个张量就变成了四个张量,也就是说所有的1都在一起了,2在一起,3在一起,4在一起,如果原张量的维度是 h * w * c ,当然这里 c 没有画出来,经过这次采样之后就得到了4个张量,每个张量的大小是 h/2、w/2,它的尺寸都缩小了一倍
- 现在把这四个张量在 c 的维度上拼接起来,也就变成了下图中所画出来的形式,张量的大小就变成了 h/2 * w/2 * 4c,相当于用空间上的维度换了更多的通道数
- 通过这个操作,就把原来一个大的张量变小了,就像卷积神经网络里的池化操作一样,为了跟卷积神经网络那边保持一致(不论是 VGGNet 还是 ResNet,一般在池化操作降维之后,通道数都会翻倍,从128变成256,从256再变成512),所以这里也只想让他翻倍,而不是变成4倍,所以紧接着又再做了一次操作,就是在 c 的维度上用一个1乘1的卷积,把通道数降下来变成2c,通过这个操作就能把原来一个大小为 h*w*c 的张量变成 h/2 * w/2 *2c 的一个张量,也就是说空间大小减半,但是通道数乘2,这样就跟卷积神经网络完全对等起来了

这里其实会发现,特征图的维度真的跟卷积神经网络好像,因为如果回想残差网络的多尺寸的特征,就是经过每个残差阶段之后的特征图大小也是56*56、28*28、14*14,最后是7*7
而且为了和卷积神经网络保持一致,Swin Transformer这篇论文并没有像 ViT 一样使用 CLS token,ViT 是给刚开始的输入序列又加了一个 CLS token,所以这个长度就从196变成了197,最后拿 CLS token 的特征直接去做分类,但 Swin Transformer 没有用这个 token,它是像卷积神经网络一样,在得到最后的特征图之后用global average polling,就是全局池化的操作,直接把7*7就取平均拉直变成1了
作者这个图里并没有画,因为 Swin Transformer的本意并不是只做分类,它还会去做检测和分割,所以说它只画了骨干网络的部分,没有去画最后的分类头或者检测头,但是如果是做分类的话,最后就变成了1*768,然后又变成了1*1,000
所以看完整个前向过程之后,就会发现 Swin Transformer 有四个 stage,还有类似于池化的 patch merging 操作,自注意力还是在小窗口之内做的以及最后还用的是 global average polling,所以说 Swin Transformer 这篇论文真的是把卷积神经网络和 Transformer 这两系列的工作完美的结合到了一起,也可以说它是披着Transformer皮的卷积神经网络
主要贡献
这篇论文的主要贡献就是基于窗口或者移动窗口的自注意力,这里作者又写了一段研究动机,就是为什么要引入窗口的自注意力,其实跟之前引言里说的都是一个事情,就是说全局自注意力的计算会导致平方倍的复杂度,同样当去做视觉里的下游任务,尤其是密集预测型的任务,或者说遇到非常大尺寸的图片时候,这种全局算自注意力的计算复杂度就非常贵了,所以就用窗口的方式去做自注意力
重点:窗口注意力
原图片会被平均的分成一些没有重叠的窗口,拿第一层之前的输入来举例,它的尺寸就是56*56*96,也就说有一个维度是56*56张量,然后把它切成一些不重叠的方格(论文中使用7*7的patch作为一个window窗口)
- 现在所有自注意力的计算都是在这些小窗口里完成的,就是说序列长度永远都是7*7=49
- 原来大的整体特征图到底里面会有多少个窗口呢?其实也就是每条边56/7就8个窗口,也就是说一共会有8*8等于64个窗口,就是说会在这64个窗口里分别去算它们的自注意力
基于窗口的自注意力模式的计算复杂度计算:

- 如果现在有一个输入,自注意力首先把它变成 q k v 三个向量,这个过程其实就是原来的向量分别乘了三个系数矩阵
- 一旦得到 query 和 k 之后,它们就会相乘,最后得到 attention,也就是自注意力的矩阵
- 有了自注意力之后,就会和 value 做一次乘法,也就相当于是做了一次加权
- 最后因为是多头自注意力,所以最后还会有一个 projection layer,这个投射层会把向量的维度投射到我们想要的维度
如果这些向量都加上它们该有的维度,也就是说刚开始输入是 h*w*c

- 公式(1)对应的是标准的多头自注意力的计算复杂度
- 每一个图片大概会有 h*w 个 patch,在刚才的例子里,h 和 w 分别都是56,c 是特征的维度
- 公式(2)对应的是基于窗口的自注意力计算的复杂度,这里的 M 就是刚才的7,也就是说一个窗口的某条边上有多少个patch
基于窗口的自注意力计算复杂度又是如何得到的呢?
- 因为在每个窗口里算的还是多头自注意力,所以可以直接套用公式(1),只不过高度和宽度变化了,现在高度和宽度不再是 h * w,而是变成窗口有多大了,也就是 M*M,也就是说现在 h 变成了 M,w 也是 M,它的序列长度只有 M * M 这么大
- 所以当把 M 值带入到公式(1)之后,就得到计算复杂度是4 * M^2 * c^2 + 2 * M^4 * c,这个就是在一个窗口里算多头自注意力所需要的计算复杂度
- 那我们现在一共有 h/M * w/M 个窗口,现在用这么多个窗口乘以每个窗口所需要的计算复杂度就能得到公式(2)了
对比公式(1)和公式(2),虽然这两个公式前面这两项是一样的,只有后面从 (h*w)^2变成了 M^2 * h * w,看起来好像差别不大,但其实如果仔细带入数字进去计算就会发现,计算复杂的差距是相当巨大的,因为这里的 h*w 如果是56*56的话, M^2 其实只有49,所以是相差了几十甚至上百倍的
这种基于窗口计算自注意力的方式虽然很好地解决了内存和计算量的问题,但是窗口和窗口之间没有通信,这样就达不到全局建模了,也就文章里说的会限制模型的能力,所以最好还是要有一种方式能让窗口和窗口之间互相通信起来,这样效果应该会更好,因为具有上下文的信息,所以作者就提出移动窗口的方式
移动窗口:
移动窗口就是把原来的窗口往右下角移动一半窗口的距离,如果Transformer是上下两层连着做这种操作,先是 window再是 shifted window 的话,就能起到窗口和窗口之间互相通信的目的了
所以说在 Swin Transformer里, transformer block 的安排是有讲究的,每次都是先要做一次基于窗口的多头自注意力,然后再做一次基于移动窗口的多头自注意力,这样就达到了窗口和窗口之间的互相通信。如下图所示

- 每次输入先进来之后先做一次 Layernorm,然后做窗口的多头自注意力,然后再过 Layernorm 过 MLP,第一个 block 就结束了
- 这个 block 结束以后,紧接着做一次Shifted window,也就是基于移动窗口的多头自注意力,然后再过 MLP 得到输出
- 这两个 block 加起来其实才算是 Swin Transformer 一个基本的计算单元,这也就是为什么stage1、2、3、4中的 swin transformer block 为什么是 *2、*2、*6、*2,也就是一共有多少层 Swin Transformer block 的数字总是偶数,因为它始终都需要两层 block连在一起作为一个基本单元,所以一定是2的倍数
到此,Swin Transformer整体的故事和结构就已经讲完了,主要的研究动机就是想要有一个层级式的 Transformer,为了这个层级式,所以介绍了 Patch Merging 的操作,从而能像卷积神经网络一样把 Transformer 分成几个阶段,为了减少计算复杂度,争取能做视觉里密集预测的任务,所以又提出了基于窗口和移动窗口的自注意力方式,也就是连在一起的两个Transformer block,最后把这些部分加在一起,就是 Swin Transformer 的结构
提高移动窗口的计算效率:
- 一个是怎样提高移动窗口的计算效率,他们采取了一种非常巧妙的 masking(掩码)的方式
- 另外一个点就是这篇论文里没有用绝对的位置编码,而是用相对的位置编码
masking(掩码)的方式计算移动窗口自注意力:为什么需要使用?
为了提高计算效率,因为如果直接计算右下图的九个窗口的自注意力,不同大小的窗口无法合并成一个batch进行计算。

- 上图是一个基础版本的移动窗口,就是把左边的窗口模式变成了右边的窗口方式
- 虽然这种方式已经能够达到窗口和窗口之间的互相通信了,但是会发现一个问题,就是原来计算的时候,特征图上只有四个窗口,但是做完移动窗口操作之后得到了9个窗口,窗口的数量增加了,而且每个窗口里的元素大小不一,比如说中间的窗口还是4*4,有16个 patch,但是别的窗口有的有4个 patch,有的有8个 patch,都不一样了,如果想做快速运算,就是把这些窗口全都压成一个 patch直接去算自注意力,就做不到了,因为窗口的大小不一样
- 有一个简单粗暴的解决方式就是把这些小窗口周围再 pad 上0 ,把它照样pad成和中间窗口一样大的窗口,这样就有9个完全一样大的窗口,这样就还能把它们压成一个batch,就会快很多
- 但是这样的话,无形之中计算复杂度就提升了,因为原来如果算基于窗口的自注意力只用算4个窗口,但是现在需要去算9个窗口,复杂度一下提升了两倍多,所以还是相当可观的
- 那怎么能让第二次移位完的窗口数量还是保持4个,而且每个窗口里的patch数量也还保持一致呢?作者提出了一个非常巧妙的掩码方式,如下图所示

上图是说,当通过普通的移动窗口方式,得到9个窗口之后,现在不在这9个窗口上算自注意力,先再做一次循环移位( cyclic shift )
- 经过这次循环移位之后,原来的窗口(虚线)就变成了现在窗口(实线)的样子,那如果在大的特征图上再把它分成四宫格的话,我在就又得到了四个窗口,意思就是说移位之前的窗口数也是4个,移完位之后再做一次循环移位得到窗口数还是4个,这样窗口的数量就固定了,也就说计算复杂度就固定了
- 但是新的问题就来了,虽然对于移位后左上角的窗口(也就是移位前最中间的窗口)来说,里面的元素都是互相紧挨着的,他们之间可以互相两两做自注意力,但是对于剩下几个窗口来说,它们里面的元素是从别的很远的地方搬过来的,所以他们之间,按道理来说是不应该去做自注意力,也就是说他们之间不应该有什么太大的联系
- 解决这个问题就需要一个很常规的操作,也就是掩码操作,这在Transformer过去的工作里是层出不穷,很多工作里都有各式各样的掩码操作
- 在 Swin Transformer这篇论文里,作者也巧妙的设计了几种掩码的方式,从而能让一个窗口之中不同的区域之间也能用一次前向过程,就能把自注意力算出来,但是互相之间都不干扰,也就是后面的 masked Multi-head Self Attention(MSA)
- 算完了多头自注意力之后,还有最后一步就是需要把循环位移再还原回去,也就是说需要把A、B、C再还原到原来的位置上去,原因是还需要保持原来图片的相对位置大概是不变的,整体图片的语义信息也是不变的,如果不把循环位移还原的话,那相当于在做Transformer的操作之中,一直在把图片往右下角移,不停的往右下角移,这样图片的语义信息很有可能就被破坏掉了
- 所以说整体而言,上图介绍了一种高效的、批次的计算方式,比如说本来移动窗口之后得到了9个窗口,而且窗口之间的patch数量每个都不一样,为了达到高效性,为了能够进行批次处理,先进行一次循环位移,把9个窗口变成4个窗口,然后用巧妙的掩码方式让每个窗口之间能够合理地计算自注意力,最后再把算好的自注意力还原,就完成了基于移动窗口的自注意力计算
掩码操作如何实现 :
作者通过这种巧妙的循环位移的方式和巧妙设计的掩码模板,从而实现了只需要一次前向过程,就能把所有需要的自注意力值都算出来,而且只需要计算4个窗口,也就是说窗口的数量没有增加,计算复杂度也没有增加,非常高效的完成了这个任务

作者给出了不同窗口的不同掩码矩阵:
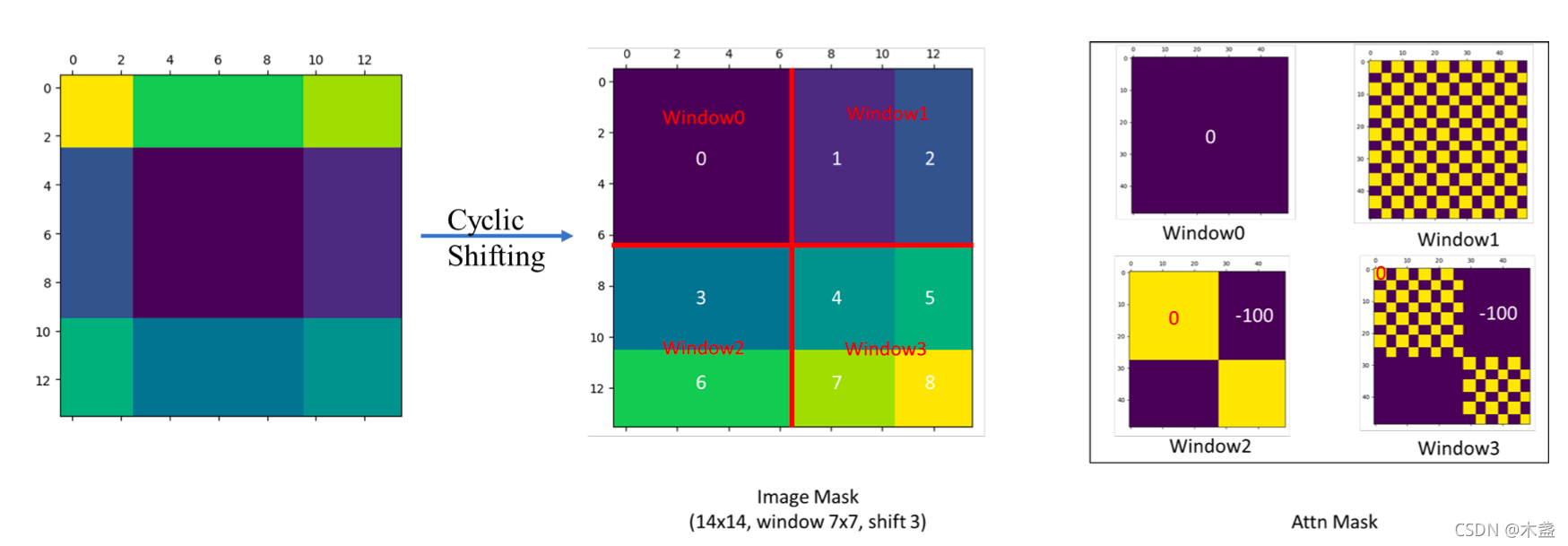
上图示例的Cyclic Shifting方法,可以保持面向计算的window数量保持不变(还是2X2),在window内部通过attention mask来计算子window中的自注意力。
Swin Transformer的几个变体
- Swin Tiny
- Swin Small
- Swin Base
- Swin Large
Swin Tiny的计算复杂度跟 ResNet-50 差不多,Swin Small 的复杂度跟 ResNet-101 是差不多的,这样主要是想去做一个比较公平的对比
这些变体之间有哪些不一样呢?,其实主要不一样的就是两个超参数
- 一个是向量维度的大小 c
- 另一个是每个 stage 里到底有多少个 transform block
这里其实就跟残差网络就非常像了,残差网络也是分成了四个 stage,每个 stage 有不同数量的残差块
实验
分类
首先是分类上的实验,这里一共说了两种预训练的方式
- 第一种就是在正规的ImageNet-1K(128万张图片、1000个类)上做预训练
- 第二种方式是在更大的ImageNet-22K(1,400万张图片、2万多个类别)上做预训练
当然不论是用ImageNet-1K去做预训练,还是用ImageNet-22K去做预训练,最后测试的结果都是在ImageNet-1K的测试集上去做的,结果如下表所示

- 上半部分是ImageNet-1K预训练的模型结果
- 下半部分是先用ImageNet-22K去预训练,然后又在ImageNet-1K上做微调,最后得到的结果
- 在表格的上半部分,作者先是跟之前最好的卷积神经网络做了一下对比,RegNet 是之前 facebook 用 NASA 搜出来的模型,EfficientNet 是 google 用NASA 搜出来的模型,这两个都算之前表现非常好的模型了,他们的性能最高会到 84.3
- 接下来作者就写了一下之前的 Vision Transformer 会达到什么效果,对于 ViT 来说,因为它没有用很好的数据增强,而且缺少偏置归纳,所以说它的结果是比较差的,只有70多
- 换上 DeiT 之后,因为用了更好的数据增强和模型蒸馏,所以说 DeiT Base 模型也能取得相当不错的结果,能到83.1
- 当然 Swin Transformer 能更高一些,Swin Base 最高能到84.5,稍微比之前最好的卷积神经网络高那么一点点,就比84.3高了0.2
- 虽然之前表现最好的 EfficientNet 的模型是在 600*600 的图片上做的,而 Swin Base 是在 384*384 的图片上做的,所以说 EfficientNet 有一些优势,但是从模型的参数和计算的 FLOPs 上来说 EfficientNet 只有66M,而且只用了 37G 的 FLOPs,但是 Swin Transformer 用了 88M 的模型参数,而且用了 47G 的 FLOPs,所以总体而言是伯仲之间
- 表格的下半部分是用 ImageNet-22k 去做预训练,然后再在ImageNet-1k上微调最后得到的结果
- 这里可以看到,一旦使用了更大规模的数据集,原始标准的 ViT 的性能也就已经上来了,对于 ViT large 来说它已经能得到 85.2 的准确度了,已经相当高了
- 但是 Swin Large 更高,Swin Large 最后能到87.3,这个是在不使用JFT-300M,就是特别大规模数据集上得到的结果,所以还是相当高的
目标检测


- 表2(a)中测试了在不同的算法框架下,Swin Transformer 到底比卷积神经网络要好多少,主要是想证明 Swin Transformer 是可以当做一个通用的骨干网络来使用的,所以用了 Mask R-CNN、ATSS、RepPointsV2 和SparseR-CNN,这些都是表现非常好的一些算法,在这些算法里,过去的骨干网络选用的都是 ResNet-50,现在替换成了 Swin Tiny
- Swin Tiny 的参数量和 FLOPs 跟 ResNet-50 是比较一致的,从后面的对比里也可以看出来,所以他们之间的比较是相对比较公平的
- 可以看到,Swin Tiny 对 ResNet-50 是全方位的碾压,在四个算法上都超过了它,而且超过的幅度也是比较大的
- 接下来作者又换了一个方式做测试,现在是选定一个算法,选定了Cascade Mask R-CNN 这个算法,然后换更多的不同的骨干网络,比如 DeiT-S、ResNet-50 和 ResNet-101,也分了几组,结果如上图中表2(b)所示
- 可以看出,在相似的模型参数和相似的 Flops 之下,Swin Transformer 都是比之前的骨干网络要表现好的
- 接下来作者又做了第三种测试的方式,如上图中的表2(c)所示,就是系统层面的比较,这个层面的比较就比较狂野了,就是现在追求的不是公平比较,什么方法都可以上,可以使用更多的数据,可以使用更多的数据增强,甚至可以在测试的使用 test time augmentation(TTA)的方式
- 可以看到,之前最好的方法 Copy-paste 在 COCO Validation Set上的结果是55.9,在 Test Set 上的结果是56,而这里如果跟最大的 Swin Transformer–Swin Large 比,它的结果分别能达到58和58.7,这都比之前高了两到三个点
语义分割

- 上图表3里可以看到之前的方法,一直到 DeepLab V3、ResNet 其实都用的是卷积神经网络,之前的这些方法其实都在44、45左右徘徊
- 但是紧接着 Vision Transformer 就来了,那首先就是 SETR 这篇论文,他们用了 ViT Large,所以就取得了50.3的这个结果
- Swin Transformer Large也取得了53.5的结果,就刷的更高了
- 其实作者这里也有标注,就是有两个“+”号的,意思是说这些模型是在ImageNet-22K 数据集上做预训练,所以结果才这么好
消融实验
实验结果如下图所示

- 上图中表4主要就是想说一下移动窗口以及相对位置编码到底对 Swin Transformer 有多有用
- 可以看到,如果光分类任务的话,其实不论是移动窗口,还是相对位置编码,它的提升相对于基线来说,也没有特别明显,当然在ImageNet的这个数据集上提升一个点也算是很显着了
- 但是他们更大的帮助,主要是出现在下游任务里,就是 COCO 和 ADE20K 这两个数据集上,也就是目标检测和语义分割这两个任务上
- 可以看到,用了移动窗口和相对位置编码以后,都会比之前大概高了3个点左右,提升是非常显着的,这也是合理的,因为如果现在去做这种密集型预测任务的话,就需要特征对位置信息更敏感,而且更需要周围的上下文关系,所以说通过移动窗口提供的窗口和窗口之间的互相通信,以及在每个 Transformer block都做更准确的相对位置编码,肯定是会对这类型的下游任务大有帮助的
总结
虽然前面已经说了很多 Swin Transformer 的影响力啊已经这么巨大了,但其实他的影响力远远不止于此,论文里这种对卷积神经网络,对 Transformer,还有对 MLP 这几种架构深入的理解和分析是可以给更多的研究者带来思考的,从而不仅可以在视觉领域里激发出更好的工作,而且在多模态领域里,相信它也能激发出更多更好的工作
BPR:用于实例分割的边界Patch优化(CVPR2021)
Look Closer to Segment Better: Boundary Patch Refinement for Instance Segmentation
代码链接:https://github.com/tinyalpha/BPR
后处理分割结果,效果是即插即用后处理模块当年的sota:通过将 BPR 框架应用于 PolyTransform + SegFix 基线,我们在 Cityscapes 排行榜上排名第一。
从目前的排名来说(22.09.23),排名第五,与top1相差不到2个百分点,而 BPR后处理使得PolyTransform + SegFix的效果提升了1.5个百分点。 相比于MASK-RCNN提升了4.2个百分点。


CVPR21上一篇关于实例分割的文章。对于Mask RCNN来说,其最终得到的mask分辨率太低,因此还原到原尺寸的时候,一些boundary信息就显得非常粗糙,导致预测生成的mask效果不尽如人意。而且处于boundary的pixel本身数量相比于整张image来说很少,同时本身难以做分类。现有的一些方法试图提升boundary quality,但预测mask边界这个task本身的复杂度和segmentation很接近了,因此开销较大。
因此本文作者提出了一种crop-and-refine的策略。首先通过经典的实例分割网络(如Mask RCNN)得到coarse mask。随后在mask的boundary出提取出一系列的patch,随后将这些patch送入一个Refinement Network,这个Refinement Network负责做二分类的语义分割,进而对boundary处的patch进行优化,整个后处理的优化网络称为BPR(Boundary Patch Refinement)。该网络可以解决传统Mask RCNN预测的mask的边界粗糙的问题。

本文的核心就是在Mask RCNN一类的网络给出coarse mask后,如何设计Refine Network来对这个粗糙 mask 的边界进行优化,进而得到resolution更高,boundary quality更好的mask。
给定一个coarse mask(上图a),首先需要决定这个mask的哪些部分要做refine。这里作者提出了一种sliding-window式的方法提取到boundary处的一系列patch(上图b)。具体来说,就是在mask边界处密集assign正方形的bounding box,这些box内部囊括了boundary pixel。随后,由于这些box有的overlap太大导致redundant(冗余),这里采用NMS进行过滤(上图c),以实现速度和精度的trade-off(平衡)。
随后这些survive下来的image patch(上图d)和mask patch(上图e)都resize到同一尺寸,一起喂入Refinement Network。这里作者argue说一定要喂入mask patch,因为一旦拥有mask patch的location和semantic信息,这个refinement network就不再需要学习instance-level semantic(实例类别信息,比如这个image patch属于哪个类别)了。所以,refinement network只需要学习boundary处的hard pixel,并把它们正确分类。
关于Refinement Network,其任务是为每一个提取出来的boundary patch独立地做二分类语义分割,任何的语义分割模型都可以搬过来做这个task。输入的通道数为4(RGB+mask),输出通道数为2(BG or FG),这里作者采用了HRNetV2(CVPR 2019),这种各种level feature不断做融合的网络可以maintain高分辨率的representation。通过合理的增加input size,boundary batch就可以得到比之前方法更高的resolution。

在对每个patch独立地refine以后,需要将它们reassemble(组装)到coarse mask上面。有的相邻的patch可能存在overlap的情况,最终的结果是取平均,以0.5作为阈值判断某个pixel属于前景或是背景。
Experiment
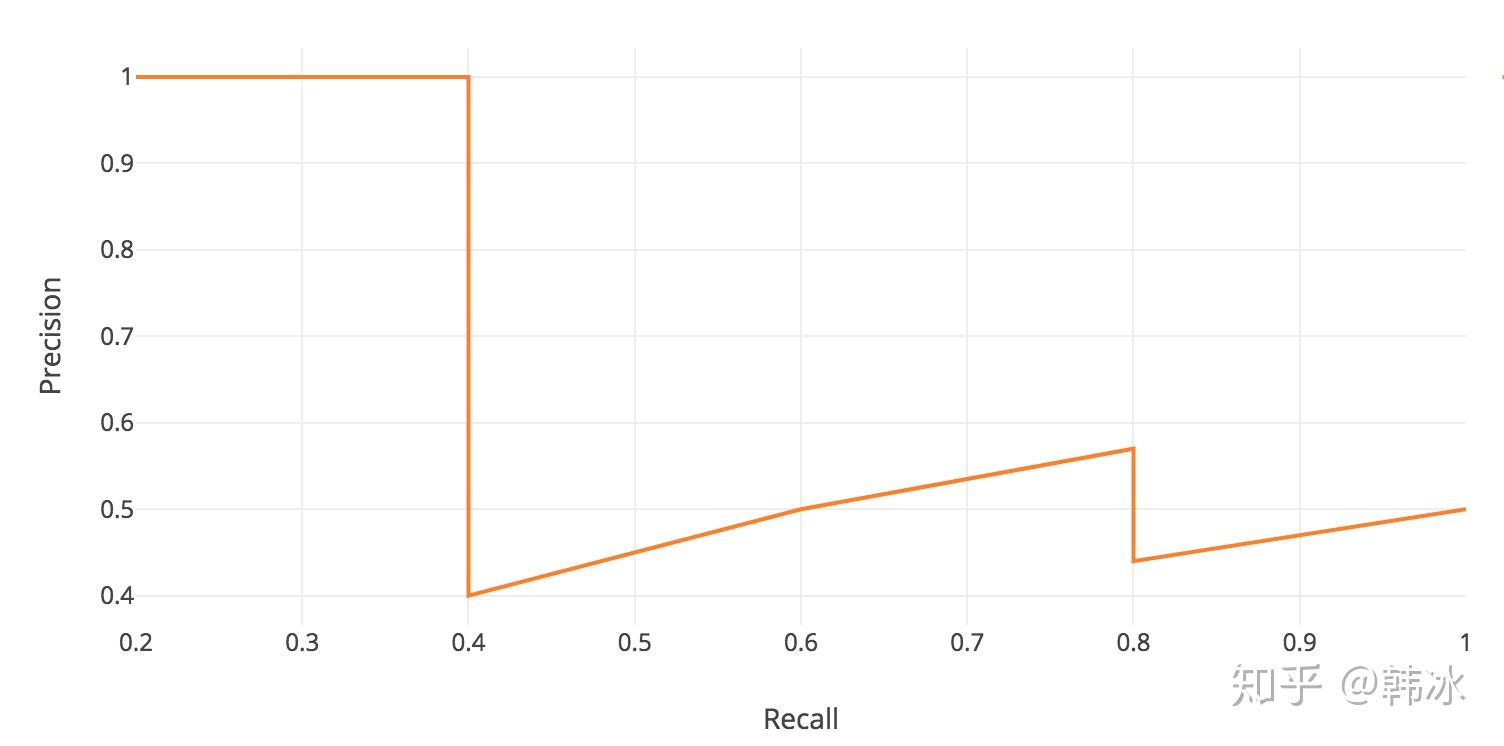
这里的指标是AP (Average precision):指的是PR曲线的面积(AP就是平均精准度,简单来说就是对PR曲线上的Precision值求均值。)对于实例分割的评价指标:使用AP评价指标
实例分割和目标检测mAP计算时除了IOU计算方式(实例分割是mask间的IOU)不同,其他都是一样的.
对于一个二分类任务,二分类器的预测结果可分为以下4类:
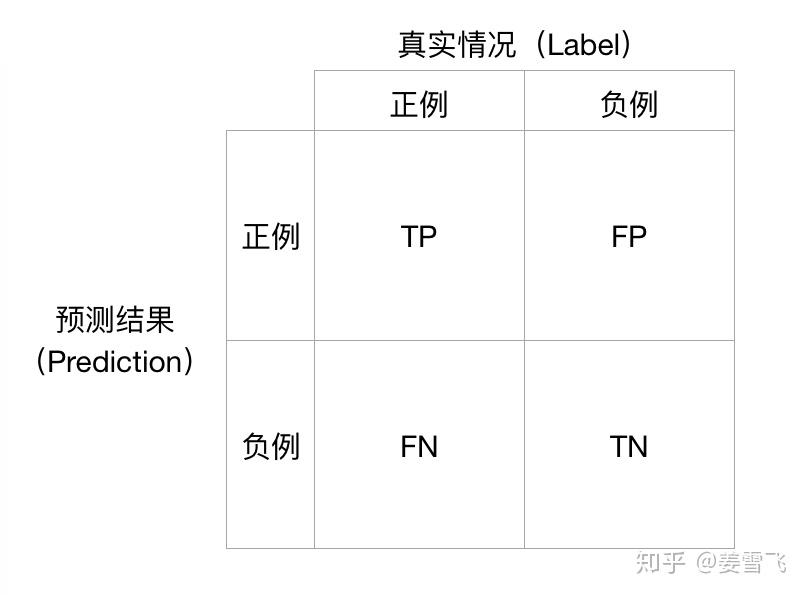
Precision的定义为:

Recall的定义为:

Precision从预测结果角度出发,描述了二分类器预测出来的正例结果中有多少是真实正例,即该二分类器预测的正例有多少是准确的;Recall从真实结果角度出发,描述了测试集中的真实正例有多少被二分类器挑选了出来,即真实的正例有多少被该二分类器召回。
逐步降低二分类器预测正例的门槛,则每次可以计算得到当前的Precision和Recall。以Recall作为横轴,Precision作为纵轴可以得到Precision-Recall曲线图,简称为P-R图。
详细解释:目标检测/实例分割中 AP 和 mAP 的混淆指标
首先通过实验证明了将mask patch一并作为输入的重要性:

patch size、不同的patch extraction策略,input size对结果的影响:


RefineNet的选取,NMS的阈值:

Cityscape上与其他方法的比较:PolyTransform + SegFix baseline,达到最高的AP。

迁移到其他model上面的结果 and coco数据集上的结果

Mask-RCNN论文

论文:http://cn.arxiv.org/pdf/1703.06870v3
代码:https://github.com/facebookresearch/maskrcnn-benchmark
Introduction
我们提出了一个简单、灵活、通用的实例分割框架,称为Mask R-CNN。我们的方法能够有效检测图像中的目标,同时为每个实例生成高质量的分割掩码。Mask R-CNN通过添加一个预测对象掩码的分支,与现有的边框识别分支并行,扩展了之前的Faster R-CNN。Mask R-CNN的训练很简单,只为Faster R-CNN增加了一小部分开销,运行速度为5帧/秒。此外,Mask R-CNN很容易泛化到其他任务,如人体姿态估计。我们展示了Mask R-CNN在COCO挑战赛的实例分割、目标检测和人物关键点检测任务上的最优结果。在不使用花哨技巧的情况下,Mask R-CNN在各项任务上都优于现有的单一模型,包括COCO 2016挑战赛的冠军。我们希望Mask R-CNN能够成为一个坚实的基线,并有助于简化未来实例识别的研究。
Fast/Faster R-CNN和Fully Convolutional Network(FCN)框架极大地推动了计算机视觉领域中目标检测和语义分割等方向的发展。这些方法的概念很直观,具有良好的灵活性和鲁棒性,并且能够快速训练和推理。我们这项工作的目标是为实例分割任务开发一个相对可行的框架。
实例分割具有一定的挑战性,因为它需要正确检测图像中的所有对象,同时还要精确分割每个实例。因此,它结合了目标检测和语义分割等计算机视觉任务中的元素。目标检测旨在对单个物体进行分类,并使用边框对每个物体进行定位。语义分割旨在将每个像素归类到一组固定的类别,而不区分对象实例。鉴于此,人们可能会认为需要一套复杂的方法才能获得良好的结果。然而,我们证明了一个令人惊讶的事实:简单、灵活、快速的系统也可以超越现有的最先进的实例分割模型。
我们的方法称为Mask R-CNN,通过在每个RoI(感兴趣区域,Region of Interest)上添加一个预测分割掩码的分支来扩展Faster R-CNN,并与现有的用于分类和边框回归的分支并行。掩码分支是应用于每个RoI的一个小FCN,以像素到像素的方式预测分割掩码,并且只会增加较小的计算开销。Mask R-CNN是基于Faster R-CNN框架而来的,易于实现和训练,有助于广泛、灵活的架构设计。
原则上,Mask R-CNN是Faster R-CNN的直观扩展,但正确构建掩码分支对于获得好的结果至关重要。最重要的是,Faster R-CNN的设计没有考虑网络输入和输出之间的像素到像素的对齐。这一点在RoIPool(处理实例的核心操作)如何执行粗空间量化来提取特征上表现得最为明显。为了修正错位,我们提出了一个简单的、没有量化的层,称为RoIAlign,它忠实地保留了精确的空间位置。尽管这看起来是一个很小的变化,但是RoIAlign有很大的影响:它将掩码精度提高了10%-50%,在更严格的localization指标下显示出更大的收益。其次,我们发现有必要将掩码和类别预测解耦:我们为每个类别独立预测一个二进制掩码,类别之间没有竞争,并依靠网络的RoI分类分支来预测类别。相比之下,FCN通常执行逐像素的多分类操作,将分割和分类耦合在一起,我们的实验结果表明这种方法的实例分割效果不佳。
在不使用花哨技巧的情况下,Mask R-CNN在COCO实例分割任务上就超越了之前的所有SOTA单模型,包括COCO 2016比赛的冠军。作为副产品,我们的方法在COCO目标检测任务上也表现出色。在消融实验中,我们评估了多个基本实例,这使我们能够证明Mask R-CNN的鲁棒性,并分析其核心因素的影响。
我们的模型可以在GPU上以每帧约200ms的速度运行,在一台8-GPU的机器上进行COCO训练需要1-2天。我们相信,快速的训练和测试,以及框架的灵活性和准确性,将有利于未来实例分割的研究。
最后,我们通过COCO关键点数据集上的人体姿态估计任务展示了Mask R-CNN框架的通用性。通过将每个关键点视为一个独热二进制掩码,只需对Mask R-CNN稍加修改,即可用于检测特定实例的姿态。Mask R-CNN超越了COCO 2016关键点检测比赛的冠军,并且能够以5帧/秒的速度运行。因此,Mask R-CNN可以被更广泛地视为一个实例识别的灵活框架,并且很容易泛化到其他更复杂的任务上。
模型方法
Mask R-CNN方法很简单:Faster R-CNN对每个候选对象有两个输出,一个是类别标签,另一个是边框偏移量。在此基础上,我们添加了第三个分支,用于输出分割掩码。因此,Mask R-CNN是一个自然且直观的想法。但是掩码输出不同于类别和边框输出,需要提取更精细的对象空间布局。接下来,我们介绍了Mask R-CNN的关键元素,包括像素到像素对齐,这是Fast/Faster R-CNN所缺失的部分。


RoIAlign:虚线网格表示特征映射图,实线边框表示RoI(Region of Interest),点表示每个边框中的4个采样点。RoIAlign通过双线性插值从特征映射图上的相邻网格点计算每个采样点的值。
- Network Architecture: 为了表述清晰,有两种分类方法
- 使用了不同的backbone:resnet-50,resnet-101,resnext-50,resnext-101;
- 使用了不同的head Architecture:Faster RCNN使用resnet50时,从Block 4导出特征供RPN使用,这种叫做ResNet-50-C4
- 作者使用除了使用上述这些结构外,还使用了一种更加高效的backbone:FPN(特征金字塔网络)

- Mask R-CNN基本结构:与Faster RCNN采用了相同的two-state结构:首先是通过一阶段网络找出RPN,然后对RPN找到的每个RoI进行分类、定位、并找到binary mask。这与当时其他先找到mask然后在进行分类的网络是不同的。
- Mask R-CNN的损失函数:
(当然了,你可以在这里调权以实现更好的效果)
- Mask的表现形式(Mask Representation):因为没有采用全连接层并且使用了RoIAlign,我们最终是在一个小feature map上做分割。
- RoIAlign:RoIPool的目的是为了从RPN网络确定的ROI中导出较小的特征图(a small feature map,eg 7×7),ROI的大小各不相同,但是RoIPool后都变成了7×7大小。RPN网络会提出若干RoI的坐标以[x,y,w,h]表示,然后输入RoI Pooling,输出7×7大小的特征图供分类和定位使用。问题就出在RoI Pooling的输出大小是7×7上,如果RON网络输出的RoI大小是8*8的,那么无法保证输入像素和输出像素是一一对应,首先他们包含的信息量不同(有的是1对1,有的是1对2),其次他们的坐标无法和输入对应起来。这对分类没什么影响,但是对分割却影响很大。RoIAlign的输出坐标使用插值算法得到,不再是简单的量化;每个grid中的值也不再使用max,同样使用差值算法。
Implementation Details
使用Fast/Faster相同的超参数,同样适用于Mask RCNN
- Training:
1、与之前相同,当IoU与Ground Truth的IoU大于0.5时才会被认为有效的RoI,只把有效RoI计算进去。
2、采用image-centric training,图像短边resize到800,每个GPU的mini-batch设置为2,每个图像生成N个RoI,在使用ResNet-50-C4 作为backbone时,N=64,在使用FPN作为backbone时,N=512。作者服务器中使用了8块GPU,所以总的minibatch是16, 迭代了160k次,初始lr=0.02,在迭代到120k次时,将lr设定到 lr=0.002,另外学习率的weight_decay=0.0001 momentum = 0.9。如果是resnext,初始lr=0.01,每个GPU的mini-batch是1。
3、RPN的anchors有5种scale,3种ratios。为了方便剥离、如果没有特别指出,则RPN网络是单独训练的且不与Mask R-CNN共享权重。但是在本论文中,RPN和Mask R-CNN使用一个backbone,所以他们的权重是共享的。(Ablation Experiments 为了方便研究整个网络中哪个部分其的作用到底有多大,需要把各部分剥离开)
- Inference:在测试时,使用ResNet-50-C4作为 backbone情况下proposal number=300,使用FPN作为 backbone时proposal number=1000。然后在这些proposal上运行bbox预测,接着进行非极大值抑制。mask分支只应用在得分最高的100个proposal上。顺序和train是不同的,但这样做可以提高速度和精度。mask 分支对于每个roi可以预测k个类别,但是我们只要背景和前景两种,所以只用k-th mask,k是根据分类分支得到的类型。然后把k-th mask resize成roi大小,同时使用阈值分割(threshold=0.5)二值化
Experiments
Main Results
在下图中可以明显看出,FCIS的分割结果中都会出现一条竖着的线(systematic artifacts),这线主要出现在物体重的部分,作者认为这是FCIS架构的问题,无法解决的。但是在Mask RCNN中没有出现。

Ablation Experiments(剥离实验)
- Architecture:
从table 2a中看出,Mask RCNN随着增加网络的深度、采用更先进的网络,都可以提高效果。注意:并不是所有的网络都是这样。 - Multinomial vs. Independent Masks:(mask分支是否进行类别预测)从table 2b中可以看出,使用sigmoid(二分类)和使用softmax(多类别分类)的AP相差很大,证明了分离类别和mask的预测是很有必要的
- Class-Specific vs. Class-Agnostic Masks:目前使用的mask rcnn都使用class-specific masks,即每个类别都会预测出一个mxm的mask,然后根据类别选取对应的类别的mask。但是使用Class-Agnostic Masks,即分割网络只输出一个mxm的mask,可以取得相似的成绩29.7vs30.3
- RoIAlign:tabel 2c证明了RoIAlign的性能
- Mask Branch:tabel 2e,FCN比MLP性能更好
Bounding Box Detection Results

- Mask RCNN精度高于Faster RCNN
- Faster RCNN使用RoI Align的精度更高
- Mask RCNN的分割任务得分与定位任务得分相近,说明Mask RCNN已经缩小了这部分差距。
Timing
- Inference:195ms一张图片,显卡Nvidia Tesla M40。其实还有速度提升的空间,比如减少proposal的数量等。
- Training:ResNet-50-FPN on COCO trainval35k takes 32 hours in our synchronized 8-GPU implementation (0.72s per 16-image mini-batch),and 44 hours with ResNet-101-FPN。
Mask R-CNN for Human Pose Estimation
让Mask R-CNN预测k个masks,每个mask对应一个关键点的类型,比如左肩、右肘,可以理解为one-hot形式。
- 使用cross entropy loss,可以鼓励网络只检测一个关键点;
- ResNet-FPN结构
- 训练了90k次,最开始lr=0.02,在迭代60k次时,lr=0.002,80k次时变为0.0002
MICCAI 2022:基于 MLP 的快速医学图像分割网络 UNeXt
论文地址: https://arxiv.org/abs/2203.04967
github:https://github.com/jeya-maria-jose/UNeXt-pytorch

Datasets
MICCAI 2022:基于 MLP 的快速医学图像分割网络 UNeXt
前言
最近 MICCAI 2022 的论文集开放下载了,地址:https://link.springer.com/book/10.1007/978-3-031-16443-9 ,每个部分的内容如下所示:
Part I: Brain development and atlases; DWI and tractography; functional brain networks; neuroimaging; heart and lung imaging; dermatology;
Part II: Computational (integrative) pathology; computational anatomy and physiology; ophthalmology; fetal imaging;
Part III: Breast imaging; colonoscopy; computer aided diagnosis;
Part IV: Microscopic image analysis; positron emission tomography; ultrasound imaging; video data analysis; image segmentation I;
Part V: Image segmentation II; integration of imaging with non-imaging biomarkers;
Part VI: Image registration; image reconstruction;
Part VII: Image-Guided interventions and surgery; outcome and disease prediction; surgical data science; surgical planning and simulation; machine learning – domain adaptation and generalization;
Part VIII: Machine learning – weakly-supervised learning; machine learning – model interpretation; machine learning – uncertainty; machine learning theory and methodologies.其中关于分割有两个部分,Image segmentation I 在 Part IV, 而 Image segmentation II 在 Part V。
随着医学图像的解决方案变得越来越适用,我们更需要关注使深度网络轻量级、快速且高效的方法。具有高推理速度的轻量级网络可以被部署在手机等设备上,例如 POCUS(point-of-care ultrasound)被用于检测和诊断皮肤状况。这就是 UNeXt 的动机。
方法概述
之前我们解读过基于 Transformer 的 U-Net 变体,近年来一直是领先的医学图像分割方法,但是参数量往往不乐观,计算复杂,推理缓慢。这篇文章提出了基于卷积多层感知器(MLP)改进 U 型架构的方法,可以用于图像分割。设计了一个 tokenized MLP 块有效地标记和投影卷积特征,使用 MLPs 来建模表示。这个结构被应用到 U 型架构的下两层中(这里我们假设纵向一共五层)。文章中提到,为了进一步提高性能,建议在输入到 MLP 的过程中改变输入的通道,以便专注于学习局部依赖关系特征。还有额外的设计就是跳跃连接了,并不是我们主要关注的地方。最终,UNeXt 将参数数量减少了 72 倍,计算复杂度降低了 68 倍,推理速度提高了 10 倍,同时还获得了更好的分割性能,如下图所示。

UNeXt 架构
UNeXt 的设计如下图所示。纵向来看,一共有两个阶段,普通的卷积和 Tokenized MLP 阶段。其中,编码器和解码器分别设计两个 Tokenized MLP 块。每个编码器将分辨率降低两倍,解码器工作相反,还有跳跃连接结构。每个块的通道数(C1-C5)被设计成超参数为了找到不掉点情况下最小参数量的网络,对于使用 UNeXt 架构的实验,遵循 C1 = 32、C2 = 64、C3 = 128、C4 = 160 和 C5 = 256。
TokMLP 设计思路
关于 Convolutional Stage 我们不做过多介绍了,在这一部分重点专注 Tokenized MLP Stage。从上一部分的图中,可以看到 Shifted MLP 这一操作,其实思路类似于 Swin transformer,引入基于窗口的注意力机制,向全局模型中添加更多的局域性。下图的意思是,Tokenized MLP 块有 2 个 MLP,在一个 MLP 中跨越宽度移动特征,在另一个 MLP 中跨越高度移动特征,也就是说,特征在高度和宽度上依次移位。论文中是这么说的:“我们将特征分成 h 个不同的分区,并根据指定的轴线将它们移到 j=5 的位置”。其实就是创建了随机窗口,这个图可以理解为灰色是特征块的位置,白色是移动之后的 padding。
(补充:MLP拥有大量参数,计算成本高且容易过度拟合,而且因为层之间的线性变换总是将前一层的输出作为一个整体,所以MLP在捕获输入特征图中的局部特征结构的能力较弱。通过轴向移动特征信息, Shifted MLP可以得到不同方向的信息流,这有助于捕获局部相关性。该操作使得我们采用纯MLP架构即可取得与CNN相同的感受野。)

解释过 Shifted MLP 后,我们再看另一部分:tokenized MLP block。首先,需要把特征转换为 tokens(可以理解为 Patch Embedding 的过程,感觉这个就是个普通卷积,而且作者为了保证conv后的矩阵减半,设置步幅为2,总之,有些编故事的意思了)。为了实现 tokenized 化,使用 kernel size 为 3 的卷积(patch_size=3, stride=2),这样会使得矩阵H和W减半,并将通道的数量改为 E,E 是 embadding 嵌入维度( token 的数量),也是一个超参数。然后把这些 token 送到上面提到的第一个跨越宽度的 MLP 中。
这里会产生了一个疑问,关于 kernel size 为 3 的卷积,使用的是什么样的卷积层?答:这里还是普通的卷积,文章中提到了 DWConv(DepthWise Conv),是后面的特征通过 DW-Conv 传递。使用 DWConv 有两个原因:(1)它有助于对 MLP 特征的位置信息进行编码。MLP 块中的卷积层足以编码位置信息,它实际上比标准的位置编码表现得更好。像 ViT 中的位置编码技术,当测试和训练的分辨率不一样时,需要进行插值,往往会导致性能下降。(2)DWConv 使用的参数数量较少。
这时我们得到了 DW-Conv 传递过来的特征,然后使用 GELU 完成激活。接下来,通过另一个 MLP(跨越height)传递特征,该 MLP 把进一步改变了特征尺寸。在这里还使用一个残差连接,将原始 token 添加为残差。然后我们利用 Layer Norm(LN),将输出特征传递到下一个块。LN 比 BN 更可取,因为它是沿着 token 进行规范化,而不是在 Tokenized MLP 块的整个批处理中进行规范化。上面这些就是一个 tokenized MLP block 的设计思路。
此外,文章中给出了 tokenized MLP block 涉及的计算公式:

其中 T 表示 tokens,H 表示高度,W 表示宽度。值得注意的是,所有这些计算都是在 embedding 维度 H 上进行的,它明显小于特征图的维度 HN×HN,其中 N 取决于 block 大小。在下面的实验部分,文章将 H 设置为 768。
实验部分
实验在 ISIC 和 BUSI 数据集上进行,可以看到,在 GLOPs、性能和推理时间都上表现不错。

下面是可视化和消融实验的部分。可视化图可以发现,UNeXt 处理的更加圆滑和接近真实标签。
消融实验可以发现,从原始的 UNet 开始,然后只是减少过滤器的数量,发现性能下降,但参数并没有减少太多。接下来,仅使用 3 层深度架构,既 UNeXt 的 Conv 阶段。显着减少了参数的数量和复杂性,但性能降低了 4%。加入 tokenized MLP block 后,它显着提高了性能,同时将复杂度和参数量是一个最小值。接下来,我们将 DWConv 添加到 positional embedding,性能又提高了。接下来,在 MLP 中添加 Shifted 操作,表明在标记化之前移位特征可以提高性能,但是不会增加任何参数或复杂性。注意:Shifted MLP 不会增加 GLOPs。


一些理解和总结
在这项工作中,提出了一种新的深度网络架构 UNeXt,用于医疗图像分割,专注于参数量的减小。UNeXt 是一种基于卷积和 MLP 的架构,其中有一个初始的 Conv 阶段,然后是深层空间中的 MLP。具体来说,提出了一个带有移位 MLP 的标记化 MLP 块。在多个数据集上验证了 UNeXt,实现了更快的推理、更低的复杂性和更少的参数数量,同时还实现了最先进的性能。
另外,个人觉得 带有移位 MLP 的标记化 MLP 块这里其实有点讲故事的意思了。
我在读这篇论文的时候,直接注意到了它用的数据集。我认为 UNeXt 可能只适用于这种简单的医学图像分割任务,类似的有 Optic Disc and Cup Seg,对于更复杂的,比如血管,软骨,Liver Tumor,kidney Seg 这些,可能效果达不到这么好,因为运算量被极大的减少了,每个 convolutional 阶段只有一个卷积层。MLP 魔改 U-Net 也算是一个尝试,在 Tokenized MLP block 中加入 DWConv 也是很合理的设计。
代码实现:
class shiftmlp(nn.Module):
def __init__(self, in_features, hidden_features=None, out_features=None, act_layer=nn.GELU, drop=0., shift_size=5):
super().__init__()
out_features = out_features or in_features
hidden_features = hidden_features or in_features
self.dim = in_features
self.fc1 = nn.Linear(in_features, hidden_features)
self.dwconv = DWConv(hidden_features)
self.act = act_layer()
self.fc2 = nn.Linear(hidden_features, out_features)
self.drop = nn.Dropout(drop)
self.shift_size = shift_size
self.pad = shift_size // 2
self.apply(self._init_weights)
def _init_weights(self, m):
if isinstance(m, nn.Linear):
trunc_normal_(m.weight, std=.02)
if isinstance(m, nn.Linear) and m.bias is not None:
nn.init.constant_(m.bias, 0)
elif isinstance(m, nn.LayerNorm):
nn.init.constant_(m.bias, 0)
nn.init.constant_(m.weight, 1.0)
elif isinstance(m, nn.Conv2d):
fan_out = m.kernel_size[0] * m.kernel_size[1] * m.out_channels
fan_out //= m.groups
m.weight.data.normal_(0, math.sqrt(2.0 / fan_out))
if m.bias is not None:
m.bias.data.zero_()
def forward(self, x, H, W):
# pdb.set_trace()
B, N, C = x.shape
xn = x.transpose(1, 2).view(B, C, H, W).contiguous()
#pad,方便后面的torch.chunk
xn = F.pad(xn, (self.pad, self.pad, self.pad, self.pad) , "constant", 0)
#按照dim=1维度,分成 self.shift_size(5)个块
xs = torch.chunk(xn, self.shift_size, 1)
#torch.roll(x,y,d)将x,沿着d维度,向上/下roll y个值
x_shift = [torch.roll(x_c, shift, 2) for x_c, shift in zip(xs, range(-self.pad, self.pad+1))]
x_cat = torch.cat(x_shift, 1)
#x.narrow(*dimension*, *start*, *length*) → Tensor 表示取变量x的第dimension维,从索引start开始到(start+length-1)范围的值。
x_cat = torch.narrow(x_cat, 2, self.pad, H)
x_s = torch.narrow(x_cat, 3, self.pad, W)
x_s = x_s.reshape(B,C,H*W).contiguous()
x_shift_r = x_s.transpose(1,2)
x = self.fc1(x_shift_r)
x = self.dwconv(x, H, W)
x = self.act(x)
x = self.drop(x)
xn = x.transpose(1, 2).view(B, C, H, W).contiguous()
xn = F.pad(xn, (self.pad, self.pad, self.pad, self.pad) , "constant", 0)
xs = torch.chunk(xn, self.shift_size, 1)
x_shift = [torch.roll(x_c, shift, 3) for x_c, shift in zip(xs, range(-self.pad, self.pad+1))]
x_cat = torch.cat(x_shift, 1)
x_cat = torch.narrow(x_cat, 2, self.pad, H)
x_s = torch.narrow(x_cat, 3, self.pad, W)
x_s = x_s.reshape(B,C,H*W).contiguous()
x_shift_c = x_s.transpose(1,2)
x = self.fc2(x_shift_c)
x = self.drop(x)
return xclass shiftedBlock(nn.Module):
def __init__(self, dim, num_heads, mlp_ratio=4., qkv_bias=False, qk_scale=None, drop=0., attn_drop=0.,
drop_path=0., act_layer=nn.GELU, norm_layer=nn.LayerNorm, sr_ratio=1):
super().__init__()
self.drop_path = DropPath(drop_path) if drop_path > 0. else nn.Identity()
self.norm2 = norm_layer(dim)
mlp_hidden_dim = int(dim * mlp_ratio)
self.mlp = shiftmlp(in_features=dim, hidden_features=mlp_hidden_dim, act_layer=act_layer, drop=drop)
self.apply(self._init_weights)
def _init_weights(self, m):
if isinstance(m, nn.Linear):
trunc_normal_(m.weight, std=.02)
if isinstance(m, nn.Linear) and m.bias is not None:
nn.init.constant_(m.bias, 0)
elif isinstance(m, nn.LayerNorm):
nn.init.constant_(m.bias, 0)
nn.init.constant_(m.weight, 1.0)
elif isinstance(m, nn.Conv2d):
fan_out = m.kernel_size[0] * m.kernel_size[1] * m.out_channels
fan_out //= m.groups
m.weight.data.normal_(0, math.sqrt(2.0 / fan_out))
if m.bias is not None:
m.bias.data.zero_()
def forward(self, x, H, W):
x = x + self.drop_path(self.mlp(self.norm2(x), H, W))
return xVision MLP —Swin-MLP
code:https://github.com/microsoft/Swin-Transformer
Swin MLP 代码来自 Swin Transformer 的官方实现。Swin Transformer 作者们在已有模型的基础上实现了 Swin MLP 模型,证明了 Window-based attention 对于 MLP 模型的有效性。
把张量 (B, H, W, C) 分成 window (B×H/M×W/M, M, M, C),其中M是 window_size。这一步相当于得到 B×H/M×W/M 个大小为 (M, M, C) 的 window。
def window_partition(x, window_size):
"""
Args:
x: (B, H, W, C)
window_size (int): window size
Returns:
windows: (num_windows*B, window_size, window_size, C)
"""
B, H, W, C = x.shape
x = x.view(B, H // window_size, window_size, W // window_size, window_size, C)
windows = x.permute(0, 1, 3, 2, 4, 5).contiguous().view(-1, window_size, window_size, C)
return windows
把 window (B×H/M×W/M, M, M, C) 变回张量 (B, H, W, C)。
def window_reverse(windows, window_size, H, W):
"""
Args:
windows: (num_windows*B, window_size, window_size, C)
window_size (int): Window size
H (int): Height of image
W (int): Width of image
Returns:
x: (B, H, W, C)
"""
B = int(windows.shape[0] / (H * W / window_size / window_size))
x = windows.view(B, H // window_size, W // window_size, window_size, window_size, -1)
x = x.permute(0, 1, 3, 2, 4, 5).contiguous().view(B, H, W, -1)
return x
一个 Swin MLP Block
class SwinMLPBlock(nn.Module):
r""" Swin MLP Block.
Args:
dim (int): Number of input channels.
input_resolution (tuple[int]): Input resolution.
num_heads (int): Number of attention heads.
window_size (int): Window size.
shift_size (int): Shift size for SW-MSA.
mlp_ratio (float): Ratio of mlp hidden dim to embedding dim.
drop (float, optional): Dropout rate. Default: 0.0
drop_path (float, optional): Stochastic depth rate. Default: 0.0
act_layer (nn.Module, optional): Activation layer. Default: nn.GELU
norm_layer (nn.Module, optional): Normalization layer. Default: nn.LayerNorm
"""
def __init__(self, dim, input_resolution, num_heads, window_size=7, shift_size=0,
mlp_ratio=4., drop=0., drop_path=0.,
act_layer=nn.GELU, norm_layer=nn.LayerNorm):
super().__init__()
self.dim = dim
self.input_resolution = input_resolution
self.num_heads = num_heads
self.window_size = window_size
self.shift_size = shift_size
self.mlp_ratio = mlp_ratio
if min(self.input_resolution) <= self.window_size:
# if window size is larger than input resolution, we don't partition windows
self.shift_size = 0
self.window_size = min(self.input_resolution)
assert 0 <= self.shift_size < self.window_size, "shift_size must in 0-window_size"
self.padding = [self.window_size - self.shift_size, self.shift_size,
self.window_size - self.shift_size, self.shift_size] # P_l,P_r,P_t,P_b
self.norm1 = norm_layer(dim)
# use group convolution to implement multi-head MLP
self.spatial_mlp = nn.Conv1d(self.num_heads * self.window_size ** 2,
self.num_heads * self.window_size ** 2,
kernel_size=1,
groups=self.num_heads)
self.drop_path = DropPath(drop_path) if drop_path > 0. else nn.Identity()
self.norm2 = norm_layer(dim)
mlp_hidden_dim = int(dim * mlp_ratio)
self.mlp = Mlp(in_features=dim, hidden_features=mlp_hidden_dim, act_layer=act_layer, drop=drop)
def forward(self, x):
H, W = self.input_resolution
B, L, C = x.shape
assert L == H * W, "input feature has wrong size"
shortcut = x
x = self.norm1(x)
x = x.view(B, H, W, C)
# shift
if self.shift_size > 0:
P_l, P_r, P_t, P_b = self.padding
shifted_x = F.pad(x, [0, 0, P_l, P_r, P_t, P_b], "constant", 0)
else:
shifted_x = x
_, _H, _W, _ = shifted_x.shape
# partition windows
x_windows = window_partition(shifted_x, self.window_size) # nW*B, window_size, window_size, C
x_windows = x_windows.view(-1, self.window_size * self.window_size, C) # nW*B, window_size*window_size, C
# Window/Shifted-Window Spatial MLP
x_windows_heads = x_windows.view(-1, self.window_size * self.window_size, self.num_heads, C // self.num_heads)
x_windows_heads = x_windows_heads.transpose(1, 2) # nW*B, nH, window_size*window_size, C//nH
x_windows_heads = x_windows_heads.reshape(-1, self.num_heads * self.window_size * self.window_size,
C // self.num_heads)
spatial_mlp_windows = self.spatial_mlp(x_windows_heads) # nW*B, nH*window_size*window_size, C//nH
spatial_mlp_windows = spatial_mlp_windows.view(-1, self.num_heads, self.window_size * self.window_size,
C // self.num_heads).transpose(1, 2)
spatial_mlp_windows = spatial_mlp_windows.reshape(-1, self.window_size * self.window_size, C)
# merge windows
spatial_mlp_windows = spatial_mlp_windows.reshape(-1, self.window_size, self.window_size, C)
shifted_x = window_reverse(spatial_mlp_windows, self.window_size, _H, _W) # B H' W' C
# reverse shift
if self.shift_size > 0:
P_l, P_r, P_t, P_b = self.padding
x = shifted_x[:, P_t:-P_b, P_l:-P_r, :].contiguous()
else:
x = shifted_x
x = x.view(B, H * W, C)
# FFN
x = shortcut + self.drop_path(x)
x = x + self.drop_path(self.mlp(self.norm2(x)))
return x
def extra_repr(self) -> str:
return f"dim={self.dim}, input_resolution={self.input_resolution}, num_heads={self.num_heads}, " \
f"window_size={self.window_size}, shift_size={self.shift_size}, mlp_ratio={self.mlp_ratio}"注意 F.pad(x, [0, 0, P_l, P_r, P_t, P_b], “constant”, 0) 的对象是 x,维度是 (B, H, W, C)。
padding相当于是第3维 (C 这一维) 不填充,第2维 (W 这一维) 左右分别填充 P_l, P_r,第1维 (H 这一维) 左右分别填充 P_t, P_b。
x_windows = window_partition(shifted_x, self.window_size) # nW*B, window_size, window_size, C:
这句代码把 shifted_x 分成 nW*B 个 windows,其中每个 window 的维度是 (window_size, window_size, C)。
# reverse shift
if self.shift_size > 0:
P_l, P_r, P_t, P_b = self.padding
x = shifted_x[:, P_t:-P_b, P_l:-P_r, :].contiguous()
else:
x = shifted_x
这里是如果进行了 shift 操作,则最后取得结果也应该是没有 padding 的部分,正好是 shifted_x[:, P_t:-P_b, P_l:-P_r, :]。
一个 Swin MLP Block 的 FLOPs,注意 WSA 的计算量是:
FLOPs (WSA) = (window_size * window_size)^2 * dim * number_window
def flops(self):
flops = 0
H, W = self.input_resolution
# norm1
flops += self.dim * H * W
# Window/Shifted-Window Spatial MLP
if self.shift_size > 0:
nW = (H / self.window_size + 1) * (W / self.window_size + 1)
else:
nW = H * W / self.window_size / self.window_size
flops += nW * self.dim * (self.window_size * self.window_size) * (self.window_size * self.window_size)
# mlp
flops += 2 * H * W * self.dim * self.dim * self.mlp_ratio
# norm2
flops += self.dim * H * W
return flops每个 stage 之间的 PatchMerging连接,把 resolution 变为一半,dim 变为2倍。
class PatchMerging(nn.Module):
r""" Patch Merging Layer.
Args:
input_resolution (tuple[int]): Resolution of input feature.
dim (int): Number of input channels.
norm_layer (nn.Module, optional): Normalization layer. Default: nn.LayerNorm
"""
def __init__(self, input_resolution, dim, norm_layer=nn.LayerNorm):
super().__init__()
self.input_resolution = input_resolution
self.dim = dim
self.reduction = nn.Linear(4 * dim, 2 * dim, bias=False)
self.norm = norm_layer(4 * dim)
def forward(self, x):
"""
x: B, H*W, C
"""
H, W = self.input_resolution
B, L, C = x.shape
assert L == H * W, "input feature has wrong size"
assert H % 2 == 0 and W % 2 == 0, f"x size ({H}*{W}) are not even."
x = x.view(B, H, W, C)
x0 = x[:, 0::2, 0::2, :] # B H/2 W/2 C
x1 = x[:, 1::2, 0::2, :] # B H/2 W/2 C
x2 = x[:, 0::2, 1::2, :] # B H/2 W/2 C
x3 = x[:, 1::2, 1::2, :] # B H/2 W/2 C
x = torch.cat([x0, x1, x2, x3], -1) # B H/2 W/2 4*C
x = x.view(B, -1, 4 * C) # B H/2*W/2 4*C
x = self.norm(x)
x = self.reduction(x)
return x
def flops(self):
H, W = self.input_resolution
# norm
flops = H * W * self.dim
# reduction
flops += (H // 2) * (W // 2) * 4 * self.dim * 2 * self.dim
return flops- Patch Merging 操作把相邻的 2×2 个 tokens 给合并到一起,得到的 token 的维度是4C。
Patch Merging 操作再通过一次线性变换把维度降为2C。
一个 Swin MLP Layer
class BasicLayer(nn.Module):
""" A basic Swin MLP layer for one stage.
Args:
dim (int): Number of input channels.
input_resolution (tuple[int]): Input resolution.
depth (int): Number of blocks.
num_heads (int): Number of attention heads.
window_size (int): Local window size.
mlp_ratio (float): Ratio of mlp hidden dim to embedding dim.
drop (float, optional): Dropout rate. Default: 0.0
drop_path (float | tuple[float], optional): Stochastic depth rate. Default: 0.0
norm_layer (nn.Module, optional): Normalization layer. Default: nn.LayerNorm
downsample (nn.Module | None, optional): Downsample layer at the end of the layer. Default: None
use_checkpoint (bool): Whether to use checkpointing to save memory. Default: False.
"""
def __init__(self, dim, input_resolution, depth, num_heads, window_size,
mlp_ratio=4., drop=0., drop_path=0.,
norm_layer=nn.LayerNorm, downsample=None, use_checkpoint=False):
super().__init__()
self.dim = dim
self.input_resolution = input_resolution
self.depth = depth
self.use_checkpoint = use_checkpoint
# build blocks
self.blocks = nn.ModuleList([
SwinMLPBlock(dim=dim, input_resolution=input_resolution,
num_heads=num_heads, window_size=window_size,
shift_size=0 if (i % 2 == 0) else window_size // 2,
mlp_ratio=mlp_ratio,
drop=drop,
drop_path=drop_path[i] if isinstance(drop_path, list) else drop_path,
norm_layer=norm_layer)
for i in range(depth)])
# patch merging layer
if downsample is not None:
self.downsample = downsample(input_resolution, dim=dim, norm_layer=norm_layer)
else:
self.downsample = None
def forward(self, x):
for blk in self.blocks:
if self.use_checkpoint:
x = checkpoint.checkpoint(blk, x)
else:
x = blk(x)
if self.downsample is not None:
x = self.downsample(x)
return x
def extra_repr(self) -> str:
return f"dim={self.dim}, input_resolution={self.input_resolution}, depth={self.depth}"
def flops(self):
flops = 0
for blk in self.blocks:
flops += blk.flops()
if self.downsample is not None:
flops += self.downsample.flops()
return flops- 包含 depth 个 Swin MLP Block。
注意计算 FLOPs 的方式:每个 blk 和 downsample 都自带 flops() 方法,可以直接来调用。
PatchEmbedded 操作
class PatchEmbed(nn.Module):
r""" Image to Patch Embedding
Args:
img_size (int): Image size. Default: 224.
patch_size (int): Patch token size. Default: 4.
in_chans (int): Number of input image channels. Default: 3.
embed_dim (int): Number of linear projection output channels. Default: 96.
norm_layer (nn.Module, optional): Normalization layer. Default: None
"""
def __init__(self, img_size=224, patch_size=4, in_chans=3, embed_dim=96, norm_layer=None):
super().__init__()
img_size = to_2tuple(img_size)
patch_size = to_2tuple(patch_size)
patches_resolution = [img_size[0] // patch_size[0], img_size[1] // patch_size[1]]
self.img_size = img_size
self.patch_size = patch_size
self.patches_resolution = patches_resolution
self.num_patches = patches_resolution[0] * patches_resolution[1]
self.in_chans = in_chans
self.embed_dim = embed_dim
self.proj = nn.Conv2d(in_chans, embed_dim, kernel_size=patch_size, stride=patch_size)
if norm_layer is not None:
self.norm = norm_layer(embed_dim)
else:
self.norm = None
def forward(self, x):
B, C, H, W = x.shape
# FIXME look at relaxing size constraints
assert H == self.img_size[0] and W == self.img_size[1], \
f"Input image size ({H}*{W}) doesn't match model ({self.img_size[0]}*{self.img_size[1]})."
x = self.proj(x).flatten(2).transpose(1, 2) # B Ph*Pw C
if self.norm is not None:
x = self.norm(x)
return x
def flops(self):
Ho, Wo = self.patches_resolution
flops = Ho * Wo * self.embed_dim * self.in_chans * (self.patch_size[0] * self.patch_size[1])
if self.norm is not None:
flops += Ho * Wo * self.embed_dim
return flops- 和 ViT 的 Patch Embedded 操作一样,本质上是一个 K=patch size,s=patch size 的 nn.Conv2d 操作,注意卷积 FLOPs 的计算公式即可。
SwinMLP 整体模型架构
class SwinMLP(nn.Module):
r""" Swin MLP
Args:
img_size (int | tuple(int)): Input image size. Default 224
patch_size (int | tuple(int)): Patch size. Default: 4
in_chans (int): Number of input image channels. Default: 3
num_classes (int): Number of classes for classification head. Default: 1000
embed_dim (int): Patch embedding dimension. Default: 96
depths (tuple(int)): Depth of each Swin MLP layer.
num_heads (tuple(int)): Number of attention heads in different layers.
window_size (int): Window size. Default: 7
mlp_ratio (float): Ratio of mlp hidden dim to embedding dim. Default: 4
drop_rate (float): Dropout rate. Default: 0
drop_path_rate (float): Stochastic depth rate. Default: 0.1
norm_layer (nn.Module): Normalization layer. Default: nn.LayerNorm.
ape (bool): If True, add absolute position embedding to the patch embedding. Default: False
patch_norm (bool): If True, add normalization after patch embedding. Default: True
use_checkpoint (bool): Whether to use checkpointing to save memory. Default: False
"""
def __init__(self, img_size=224, patch_size=4, in_chans=3, num_classes=1000,
embed_dim=96, depths=[2, 2, 6, 2], num_heads=[3, 6, 12, 24],
window_size=7, mlp_ratio=4., drop_rate=0., drop_path_rate=0.1,
norm_layer=nn.LayerNorm, ape=False, patch_norm=True,
use_checkpoint=False, **kwargs):
super().__init__()
self.num_classes = num_classes
self.num_layers = len(depths)
self.embed_dim = embed_dim
self.ape = ape
self.patch_norm = patch_norm
self.num_features = int(embed_dim * 2 ** (self.num_layers - 1))
self.mlp_ratio = mlp_ratio
# split image into non-overlapping patches
self.patch_embed = PatchEmbed(
img_size=img_size, patch_size=patch_size, in_chans=in_chans, embed_dim=embed_dim,
norm_layer=norm_layer if self.patch_norm else None)
num_patches = self.patch_embed.num_patches
patches_resolution = self.patch_embed.patches_resolution
self.patches_resolution = patches_resolution
# absolute position embedding
if self.ape:
self.absolute_pos_embed = nn.Parameter(torch.zeros(1, num_patches, embed_dim))
trunc_normal_(self.absolute_pos_embed, std=.02)
self.pos_drop = nn.Dropout(p=drop_rate)
# stochastic depth
dpr = [x.item() for x in torch.linspace(0, drop_path_rate, sum(depths))] # stochastic depth decay rule
# build layers
self.layers = nn.ModuleList()
for i_layer in range(self.num_layers):
layer = BasicLayer(dim=int(embed_dim * 2 ** i_layer),
input_resolution=(patches_resolution[0] // (2 ** i_layer),
patches_resolution[1] // (2 ** i_layer)),
depth=depths[i_layer],
num_heads=num_heads[i_layer],
window_size=window_size,
mlp_ratio=self.mlp_ratio,
drop=drop_rate,
drop_path=dpr[sum(depths[:i_layer]):sum(depths[:i_layer + 1])],
norm_layer=norm_layer,
downsample=PatchMerging if (i_layer < self.num_layers - 1) else None,
use_checkpoint=use_checkpoint)
self.layers.append(layer)
self.norm = norm_layer(self.num_features)
self.avgpool = nn.AdaptiveAvgPool1d(1)
self.head = nn.Linear(self.num_features, num_classes) if num_classes > 0 else nn.Identity()
self.apply(self._init_weights)
def _init_weights(self, m):
if isinstance(m, (nn.Linear, nn.Conv1d)):
trunc_normal_(m.weight, std=.02)
if m.bias is not None:
nn.init.constant_(m.bias, 0)
elif isinstance(m, nn.LayerNorm):
nn.init.constant_(m.bias, 0)
nn.init.constant_(m.weight, 1.0)
@torch.jit.ignore
def no_weight_decay(self):
return {'absolute_pos_embed'}
@torch.jit.ignore
def no_weight_decay_keywords(self):
return {'relative_position_bias_table'}
def forward_features(self, x):
x = self.patch_embed(x)
if self.ape:
x = x + self.absolute_pos_embed
x = self.pos_drop(x)
for layer in self.layers:
x = layer(x)
x = self.norm(x) # B L C
x = self.avgpool(x.transpose(1, 2)) # B C 1
x = torch.flatten(x, 1)
return x
def forward(self, x):
x = self.forward_features(x)
x = self.head(x)
return x
def flops(self):
flops = 0
flops += self.patch_embed.flops()
for i, layer in enumerate(self.layers):
flops += layer.flops()
# adaptive average pool
flops += self.num_features * self.patches_resolution[0] * self.patches_resolution[1] // (2 ** self.num_layers)
# head
flops += self.num_features * self.num_classes
return flops- 由4个 Stage 组成,每个 Stage 由 BasicLayer 实现。
传入的 depths 代表每个 Stage 的层数,比如 Swin-T 就是:[2, 2, 6, 2]。
经典CNN网络结构
回顾21世纪10年代,深度学习取得了巨大的进步,产生了巨大的影响。主要驱动力是神经网络的复兴,特别是卷积神经网络(ConvNets)。十年来,视觉识别领域成功地从工程特征转变为设计(ConvNet)架构。尽管采用反向传播训练方法的卷积神经网络自上世纪八十年代已经发明了,但直到2012年我们才看到它作为视觉特征学习的真正潜力。AlexNet的引入促成了“ImageNet的时刻”,引领了计算机视觉领域的一个新时代,这个领域因此而快速演化。具有代表性的网络有VGGNet、Inceptions、Resnet、DenseNet、MobileNet、EfficientNet、RegNet等,它们分别关注精度、效率、可扩展性等方面,并且普及了很多有用的设计原则。
1、 VGGNet
VGG16相比AlexNet的一个改进是采用连续的几个3×3的卷积核代替AlexNet中的较大卷积核(11×11,7×7,5×5)。对于给定的感受野(与输出有关的输入图片的局部大小),采用堆积的小卷积核是优于采用大的卷积核,因为多层非线性层可以增加网络深度来保证学习更复杂的模式,而且代价还比较小(参数更少)。
简单来说,在VGG中,使用了3个3×3卷积核来代替7×7卷积核,使用了2个3×3卷积核来代替5*5卷积核,这样做的主要目的是在保证具有相同感知野的条件下,提升了网络的深度,在一定程度上提升了神经网络的效果。
网络结构:

2、 Inception系列网络
Inception V1
在这之前,网络大都是这样子的:

也就是卷积层和池化层的顺序连接。这样的话,要想提高精度,增加网络深度和宽度是一个有效途径,但也面临着参数量过多、过拟合等问题。(当然,改改超参数也可以提高性能)
有没有可能在同一层就可以提取不同(稀疏或不稀疏)的特征呢(使用不同尺寸的卷积核)?于是,2014年,在其他人都还在一味的增加网络深度时(比如vgg),GoogleNet就率先提出了卷积核的并行合并(也称Bottleneck Layer),如下图。
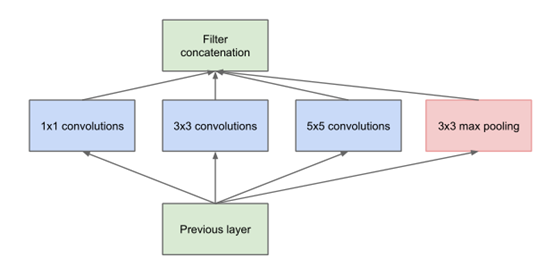
和卷积层、池化层顺序连接的结构(如VGG网络)相比,这样的结构主要有以下改进:
- 一层block就包含1×1卷积,3×3卷积,5×5卷积,3×3池化(使用这样的尺寸不是必需的,可以根据需要进行调整)。这样,网络中每一层都能学习到“稀疏”(3×3、5×5)或“不稀疏”(1×1)的特征,既增加了网络的宽度,也增加了网络对尺度的适应性;
- 通过deep concat在每个block后合成特征,获得非线性属性。
按照这样的结构来增加网络的深度,虽然可以提升性能,但是还面临计算量大(参数多)的问题。为改善这种现象,GooLeNet借鉴Network-in-Network的思想,使用1×1的卷积核实现降维操作(也间接增加了网络的深度),以此来减小网络的参数量(这里就不对两种结构的参数量进行定量比较了),如图所示。
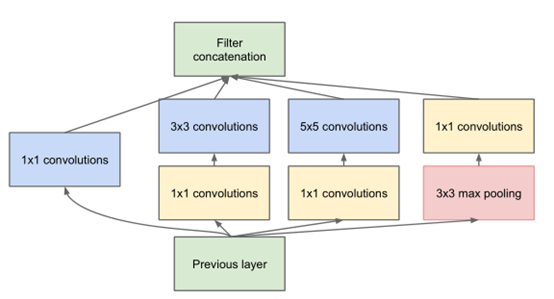
最后实现的inception v1网络是上图结构的顺序连接,其中不同inception模块之间使用2×2的最大池化进行下采样,如表所示。
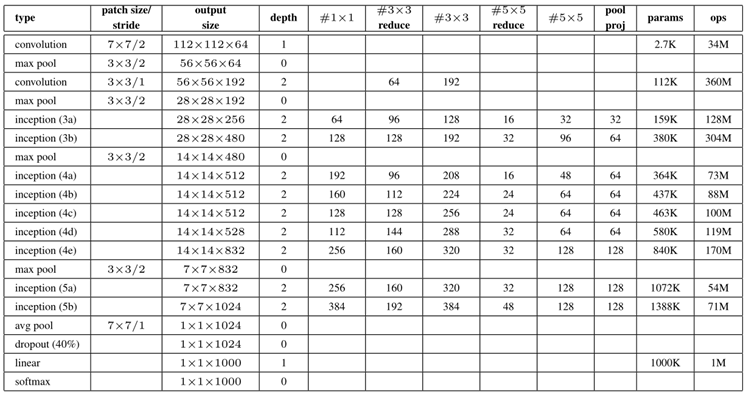
如表所示,实现的网络仍有一层全连接层,该层的设置是为了迁移学习的实现(下同)。
在之前的网络中,最后都有全连接层,经实验证明,全连接层并不是很必要的,因为可能会带来以下三点不便:
- 网络的输入需要固定
- 参数量多
- 易发生过拟合
实验证明,将其替换为平均池化层(或者1×1卷积层)不仅不影响精度,还可以减少参数量。
此外,实验室的小伙伴最近做了下实验,如果是小目标检测的话,网络的最后还是需要几层全连接层的,猜想可能是用池化的话会损失太多信息,毕竟是小目标。
———————————————————————————————–
Inception V2和Inception V3的改进,主要是基于V3论文中提到的四个原则:
- 避免表示瓶颈,尤其是在网络的前面。一般来说,特征图从输入到输出应该缓慢减小。
- 高维度特征在网络局部处理更加容易。考虑到更多的耦合特征,在卷积网络中增加非线性。可以让网络训练更快。
- 空间聚合可以以低维度嵌入进行,这样不会影响特征的表达能力。如,在进行大尺度卷积之前,先对输入进行降维。
- 平衡网络的宽度和深度。增加宽度和深度都会带来性能上的提升,两者同时增加带来了并行提升,但是要考虑计算资源的合理分配。
———————————————————————————————–
Inception v2
(注意,这里实现的inception v2的结构是在inception v3论文中有介绍)
2015年Google团队又提出了inception v2的结构,基于上面提到的一些原则,在V1的基础之上主要做了以下改进:
⑴ 使用BN层,将每一层的输出都规范化到一个N(0,1)的正态分布,这将有助于训练,因为下一层不必学习输入数据中的偏移,并且可以专注与如何更好地组合特征(也因为在v2里有较好的效果,BN层几乎是成了深度网络的必备);
(在Batch-normalized论文中只增加了BN层,而之后的Inception V3的论文提及到的inception v2还做了下面的优化)
⑵ 使用2个3×3的卷积代替梯度(特征图,下同)为35×35中的5×5的卷积,这样既可以获得相同的视野(经过2个3×3卷积得到的特征图大小等于1个5×5卷积得到的特征图),还具有更少的参数,还间接增加了网络的深度,如下图。(基于原则3)
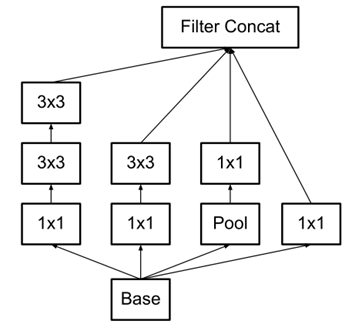
⑶ 3×3的卷积核表现的不错,那更小的卷积核是不是会更好呢?比如2×2。对此,v2在17×17的梯度中使用1*n和n*1这种非对称的卷积来代替n*n的对称卷积,既降低网络的参数,又增加了网络的深度(实验证明,该结构放于网络中部,取n=7,准确率更高),如下。(基于原则3)
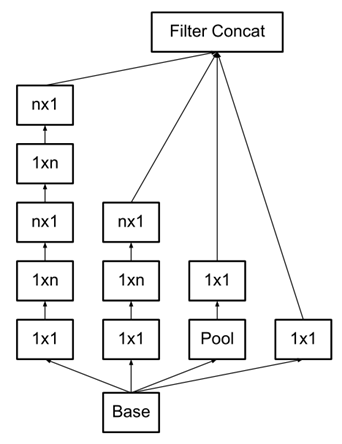
⑷ 在梯度为8×8时使用可以增加滤波器输出的模块(如下图),以此来产生高维的稀疏特征。(基于原则2)
(原则2指出,在高维特征上,采用这种结构更好,因此该模块用在了8×8的梯度上)
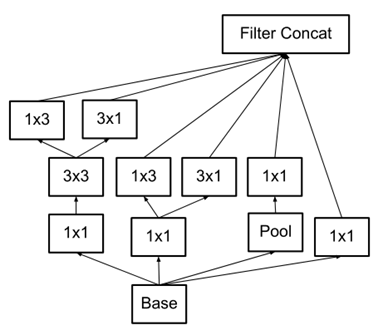
⑸ 输入从224×224变为229×229。
最后实现的Inception v2的结构如下表。
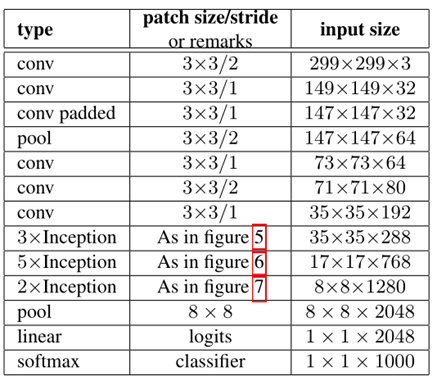
经过网络的改进,inception v2得到更低的识别误差率,与其他网络识别误差率对比如表所示。
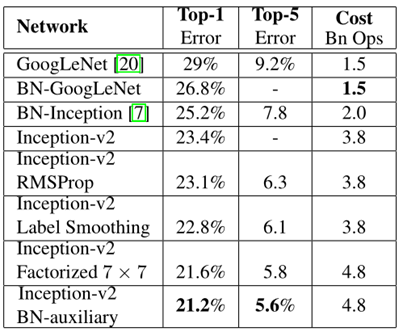
如表,inception v2相比inception v1在imagenet的数据集上,识别误差率由29%降为23.4%。
Inception v3
inception模块之间特征图的缩小,主要有下面两种方式:
右图是先进行inception操作,再进行池化来下采样,但是这样参数量明显多于左图(比较方式同前文的降维后inception模块),因此v2采用的是左图的方式,即在不同的inception之间(35/17/8的梯度)采用池化来进行下采样。
但是,左图这种操作会造成表达瓶颈问题,也就是说特征图的大小不应该出现急剧的衰减(只经过一层就骤降)。如果出现急剧缩减,将会丢失大量的信息,对模型的训练造成困难。(上文提到的原则1)
因此,在2015年12月提出的Inception V3结构借鉴inception的结构设计了采用一种并行的降维结构,如下图:
具体来说,就是在35/17/8之间分别采用下面这两种方式来实现特征图尺寸的缩小,如下图:
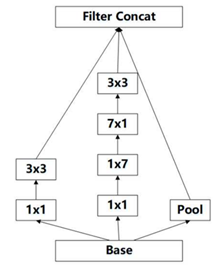
这样就得到Inception v3的网络结构,如表所示。
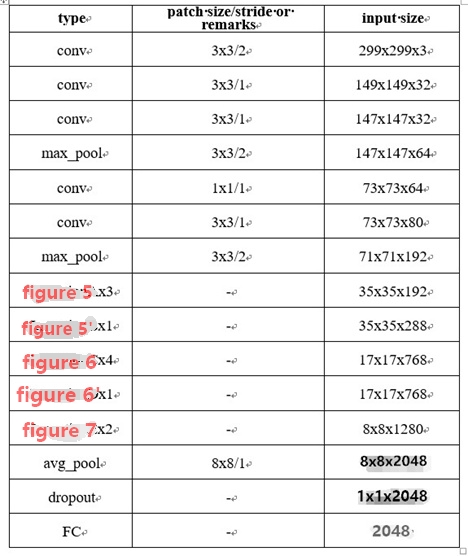
经过优化后的inception v3网络与其他网络识别误差率对比如表所示。
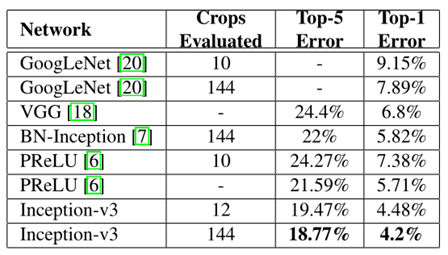
如表所示,在144×144的输入上,inception v3的识别错误率由v1的7.89%降为了4.2%。
此外,文章还提到了中间辅助层,即在网络中部再增加一个输出层。实验发现,中间辅助层在训练前期影响不大,而在训练后期却可以提高精度,相当于正则项。
Inception V4
其实,做到现在,inception模块感觉已经做的差不多了,再做下去准确率应该也不会有大的改变。但是谷歌这帮人还是不放弃,非要把一个东西做到极致,改变不了inception模块,就改变其他的。
因此,作者Christian Szegedy设计了inception v4的网络,将原来卷积、池化的顺次连接(网络的前几层)替换为stem模块,来获得更深的网络结构。stem模块结构如下
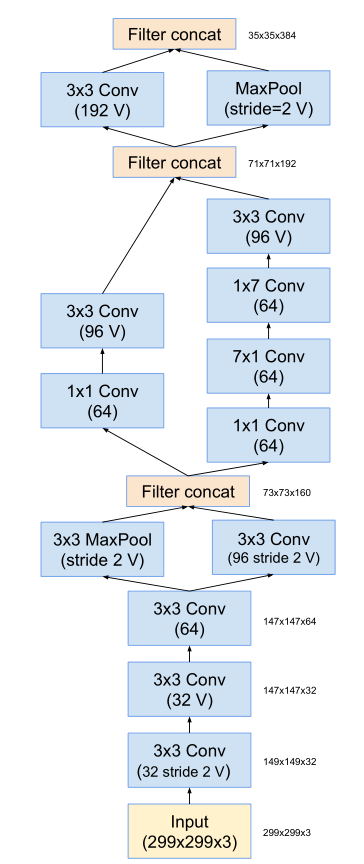
stem之后的,同v3,是inception模块和reduction模块,如下图
最终得到的inception v4结构如下图。
Inception-ResNet-v2
ResNet(该网络介绍见卷积神经网络结构简述(三)残差系列网络)的结构既可以加速训练,还可以提升性能(防止梯度弥散);Inception模块可以在同一层上获得稀疏或非稀疏的特征。有没有可能将两者进行优势互补呢?
Christian Szegedy等人将两个模块的优势进行了结合,设计出了Inception-ResNet网络。
(inception-resnet有v1和v2两个版本,v2表现更好且更复杂,这里只介绍了v2)
inception-resnet的成功,主要是它的inception-resnet模块。
inception-resnet v2中的Inception-resnet模块如下图
Inception-resnet模块之间特征图尺寸的减小如下图。(类似于inception v4)
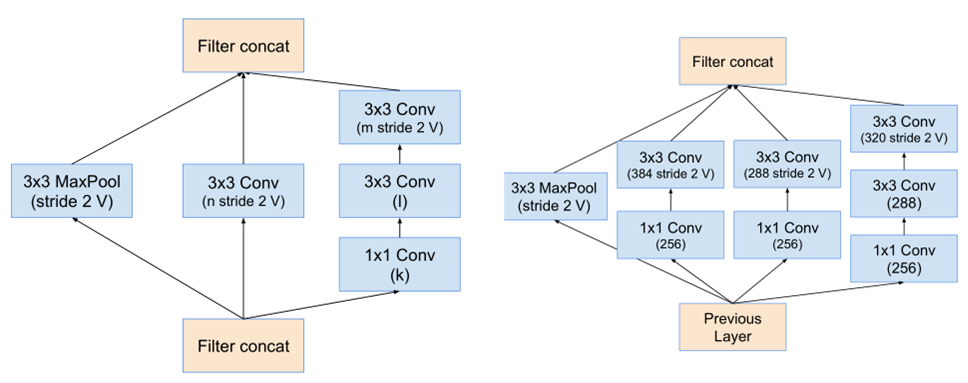
最终得到的Inception-ResNet-v2网络结构如图(stem模块同inception v4)。
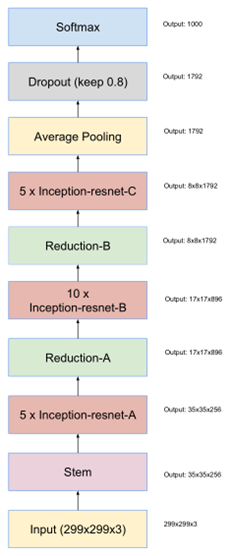
经过这两种网络的改进,使得模型对图像识别的错误率进一步得到了降低。Inception、resnet网络结果对比如表所示。
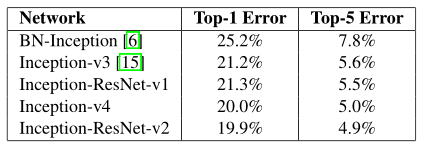
如表,Inception V4与Inception-ResNet-v2网络较之前的网络,误差率均有所下降。
3、Resnet
ResNet网络是在2015年由微软实验室提出,斩获当年ImageNet竞赛中分类任务第一名,目标检测第一名。获得COCO数据集中目标检测第一名,图像分割第一名。下图是ResNet34层模型的结构简图。
在ResNet网络中有如下几个亮点:
(1)提出residual结构(残差结构),并搭建超深的网络结构(突破1000层)
(2)使用Batch Normalization加速训练(丢弃dropout)
在ResNet网络提出之前,传统的卷积神经网络都是通过将一系列卷积层与下采样层进行堆叠得到的。但是当堆叠到一定网络深度时,就会出现两个问题。1)梯度消失或梯度爆炸。 2)退化问题(degradation problem)。在ResNet论文中说通过数据的预处理以及在网络中使用BN(Batch Normalization)层能够解决梯度消失或者梯度爆炸问题。如果不了解BN层可参考这个链接。但是对于退化问题(随着网络层数的加深,效果还会变差,如下图所示)并没有很好的解决办法。

4、DenseNet
DenseNet模型,它的基本思路与ResNet一致,但是它建立的是前面所有层与后面层的密集连接(dense connection),它的名称也是由此而来。DenseNet的另一大特色是通过特征在channel上的连接来实现特征重用(feature reuse)。这些特点让DenseNet在参数和计算成本更少的情形下实现比ResNet更优的性能,DenseNet也因此斩获CVPR 2017的最佳论文奖.
相比ResNet,DenseNet提出了一个更激进的密集连接机制:即互相连接所有的层,具体来说就是每个层都会接受其前面所有层作为其额外的输入。图1为ResNet网络的连接机制,作为对比,图2为DenseNet的密集连接机制。可以看到,ResNet是每个层与前面的某层(一般是2~3层)短路连接在一起,连接方式是通过元素级相加。而在DenseNet中,每个层都会与前面所有层在channel维度上连接(concat)在一起(这里各个层的特征图大小是相同的),并作为下一层的输入。
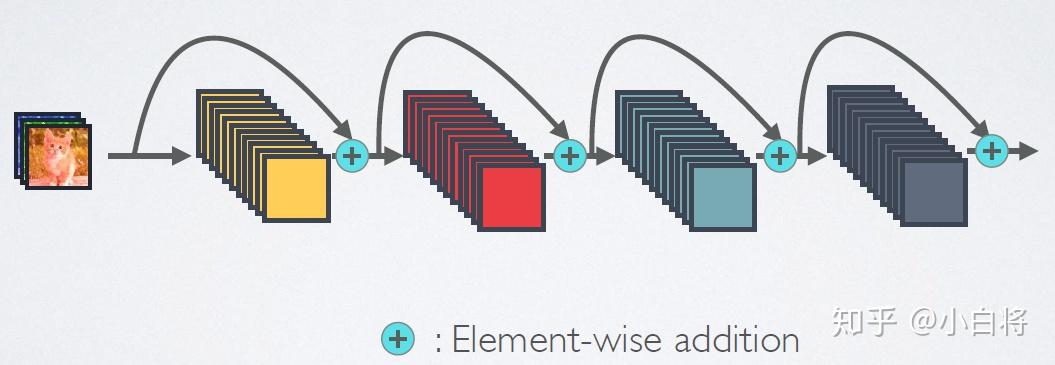

5、MobileNet
MobileNet的基本单元是深度级可分离卷积(depthwise separable convolution),其实这种结构之前已经被使用在Inception模型中。深度级可分离卷积其实是一种可分解卷积操作(factorized convolutions),其可以分解为两个更小的操作:depthwise convolution和pointwise convolution,如图1所示。Depthwise convolution和标准卷积不同,对于标准卷积其卷积核是用在所有的输入通道上(input channels),而depthwise convolution针对每个输入通道采用不同的卷积核,就是说一个卷积核对应一个输入通道,所以说depthwise convolution是depth级别的操作。而pointwise convolution其实就是普通的卷积,只不过其采用1×1的卷积核。图2中更清晰地展示了两种操作。对于depthwise separable convolution,其首先是采用depthwise convolution对不同输入通道分别进行卷积,然后采用pointwise convolution将上面的输出再进行结合,这样其实整体效果和一个标准卷积是差不多的,但是会大大减少计算量和模型参数量。
前面讲述了depthwise separable convolution,这是MobileNet的基本组件,但是在真正应用中会加入batchnorm,并使用ReLU激活函数,所以depthwise separable convolution的基本结构如图3所示。
MobileNet的网络结构如表1所示。首先是一个3×3的标准卷积,然后后面就是堆积depthwise separable convolution,并且可以看到其中的部分depthwise convolution会通过strides=2进行down sampling。然后采用average pooling将feature变成1×1,根据预测类别大小加上全连接层,最后是一个softmax层。如果单独计算depthwise
convolution和pointwise convolution,整个网络有28层(这里Avg Pool和Softmax不计算在内)。我们还可以分析整个网络的参数和计算量分布,如表2所示。可以看到整个计算量基本集中在1×1卷积上,如果你熟悉卷积底层实现的话,你应该知道卷积一般通过一种im2col方式实现,其需要内存重组,但是当卷积核为1×1时,其实就不需要这种操作了,底层可以有更快的实现。对于参数也主要集中在1×1卷积,除此之外还有就是全连接层占了一部分参数。
MobileNetv2
MobileNetv2相比v1的两个主要改进:linear bottleneck和inverted residual。
v2的加入了1×1升维,引入Shortcut并且去掉了最后的ReLU,改为Linear。步长为1时,先进行1×1卷积升维,再进行深度卷积提取特征,再通过Linear的逐点卷积降维。将input与output相加,形成残差结构。步长为2时,因为input与output的尺寸不符,因此不添加shortcut结构,其余均一致。
首先利用3×3的深度可分离卷积提取特征,然后利用1×1的卷积来扩张通道。用这样的block堆叠起来的MobileNetV1既能较少不小的参数量、计算量,提高网络运算速度,又能的得到一个接近于标准卷积的还不错的结果,看起来是很美好的。
但是!
有人在实际使用的时候, 发现深度卷积部分的卷积核比较容易训废掉:训完之后发现深度卷积训出来的卷积核有不少是空的.
这是为什么?
作者认为这是ReLU这个浓眉大眼的激活函数的锅。(没想到你个浓眉大眼的ReLU激活函数也叛变革命了???)
针对这个问题,可以这样解决:既然是ReLU导致的信息损耗,将ReLU替换成线性激活函数。我们当然不能把所有的激活层都换成线性的啊,所以我们就悄咪咪的把最后的那个ReLU6换成Linear。作者将这个部分称之为linear bottleneck。对,就是论文名中的那个linear bottleneck。
现在还有个问题是,深度卷积本身没有改变通道的能力,来的是多少通道输出就是多少通道。如果来的通道很少的话,DW深度卷积只能在低维度上工作,这样效果并不会很好,所以我们要“扩张”通道。既然我们已经知道PW逐点卷积也就是1×1卷积可以用来升维和降维,那就可以在DW深度卷积之前使用PW卷积进行升维(升维倍数为t,t=6),再在一个更高维的空间中进行卷积操作来提取特征:
也就是说,不管输入通道数是多少,经过第一个PW逐点卷积升维之后,深度卷积都是在相对的更高6倍维度上进行工作。
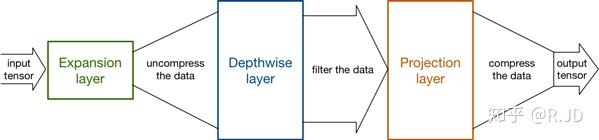
回顾V1的网络结构,我们发现V1很像是一个直筒型的VGG网络。我们想像Resnet一样复用我们的特征,所以我们引入了shortcut结构,这样V2的block就是如下图形式:
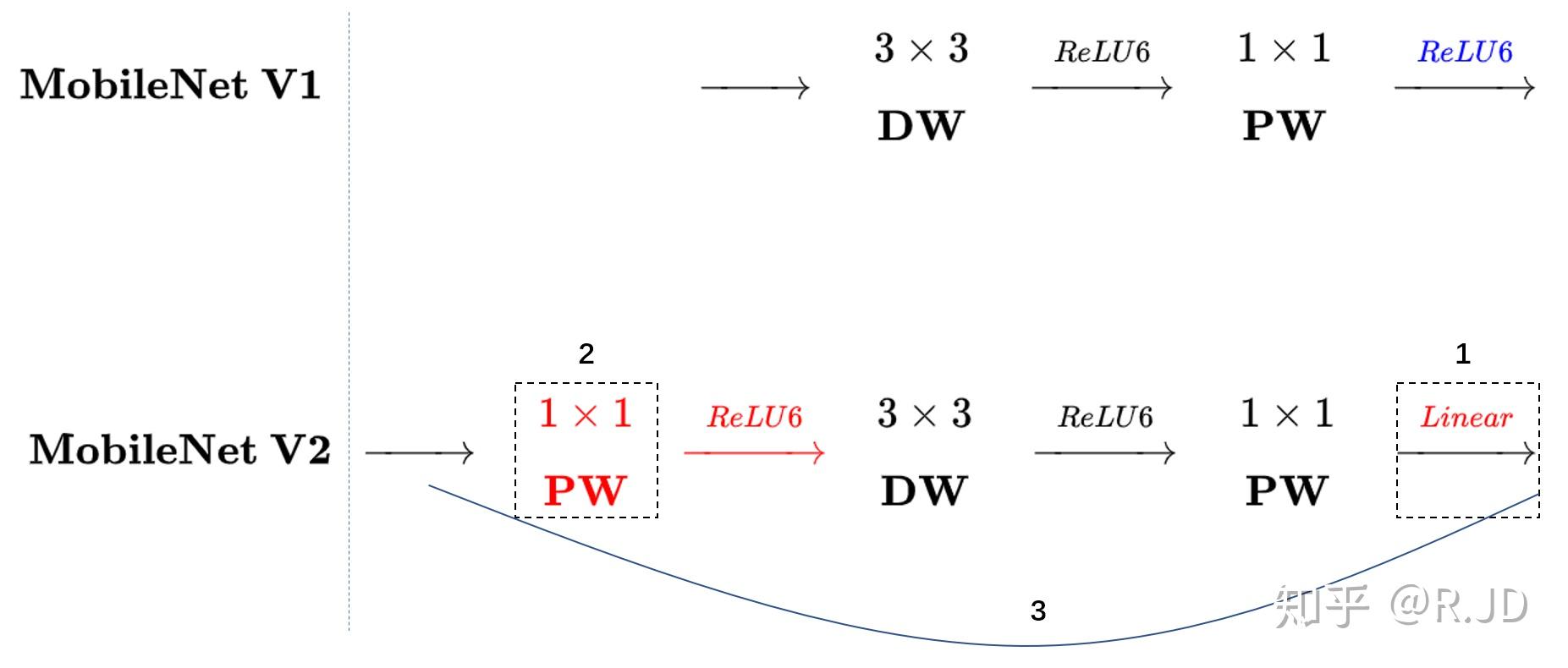
对比一下V1和V2:
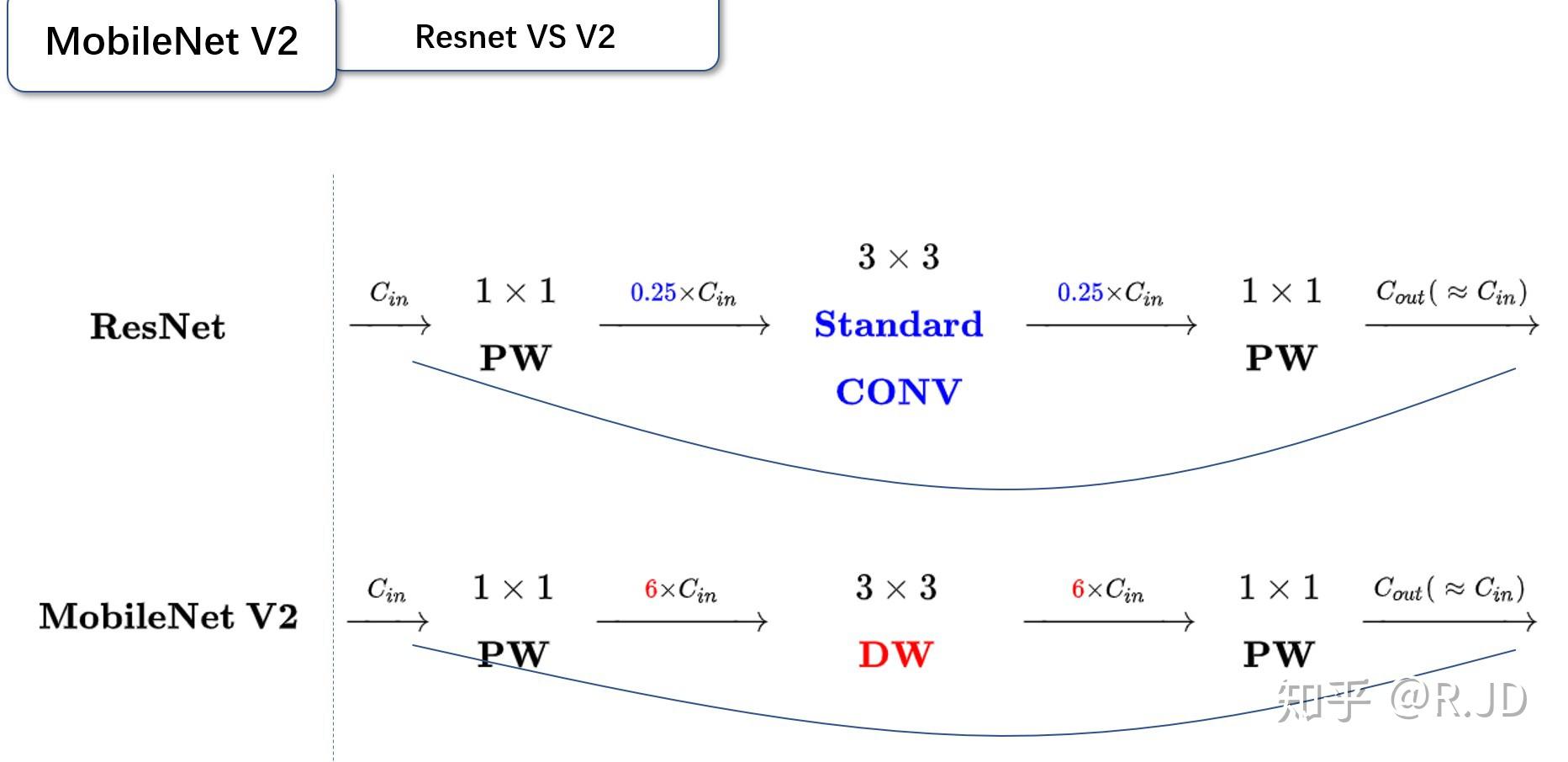
可以发现,都采用了 1×1 -> 3 ×3 -> 1 × 1 的模式,以及都使用Shortcut结构。但是不同点呢:
- ResNet 先降维 (0.25倍)、卷积、再升维。
- MobileNetV2 则是 先升维 (6倍)、卷积、再降维。
刚好V2的block刚好与Resnet的block相反,作者将其命名为Inverted residuals。就是论文名中的Inverted residuals。
6、EfficientNet
该论文提出了一种新的模型缩放方法,它使用一个简单而高效的复合系数来以更结构化的方式放大 CNNs。 不像传统的方法那样任意缩放网络维度,如宽度,深度和分辨率,该论文的方法用一系列固定的尺度缩放系数来统一缩放网络维度。 通过使用这种新颖的缩放方法和 AutoML[5] 技术,作者将这种模型称为 EfficientNets ,它具有最高达10倍的效率(更小、更快)。
模型扩展的有效性在很大程度上依赖于baseline网络。为了进一步提高性能,作者还开发了一个新的基线网络,通过使用 AutoML MNAS 框架执行神经结构搜索,优化了准确性和效率。 最终的架构使用移动反向bottleneck卷积(MBConv) ,类似于 mobileenetv2和 MnasNet。

移动翻转瓶颈卷积(mobile inverted bottleneck convolution,MBConv),类似于 MobileNetV2 和 MnasNet,由深度可分离卷积Depthwise Convolution和SENet构成。
每个MBConv的网络结构如下:
MBConv = 1×1升维 + Depthwise Convolution + SENet + 1×1降维 + add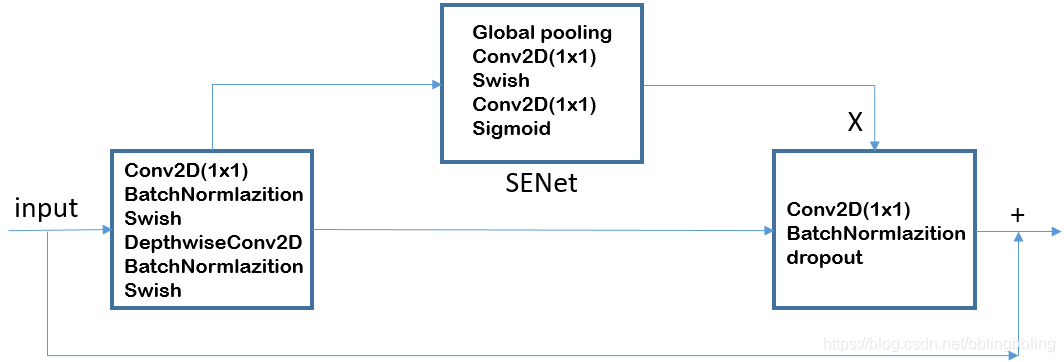
SENet
该网络为压缩与激发网络(Squeeze-and-Excitation Network,SENet),即注意力机制。该思想由Momenta公司提出,并发于2017CVPR。SENet网络的创新点在于关注channel之间的关系,希望模型可以自动学习到不同channel特征的重要程度。
其中第一个FC层降维,降维系数为r,然后ReLU激活,最后的FC层恢复原始的维度。
SENet添加位置示意:
代码实现
7、RegNet
本文的基本贡献有三方面。
- 提出了设计空间的设计原则。
- 根据这些原则,一个有效的设计空间被引入(RegNet)
- 介绍了一组SoTA网络(RegNetX和RegNetY)。
我们首先设计了一个 AnyNet,它包含三个部分
- Stem 一个简单的网络输入头
- body 网络中主要的运算量都在这里
- head 用于预测分类的输出头
我们将 stem 和 head 固定下来,并专注于网络 body 设计。因为 body 部分的参数量最多,运算量也多,这部分是决定网络准确性的关键
而 Body 结构,通常包含 4 个 stage,每个 stage 都会进行降采样。而 1 个 stage 是由多个 block 进行堆叠得到的。
论文中,我们的 Block 采取的是带有组卷积的残差 BottleNeck Block(即 ResNext 里的结构),我们称在这样 Block 限制条件下的搜索空间为 AnyNetX ,Block 的结构如下:
此时 AnyNetX 中有 16 个自由度可以设计,包含了 4 个 stage,每个 stage 有 4 个 Block 参数:
- block 的数目 di
- block 的宽度 wi
- Bottleneck 的通道缩放比例 bi
- 分组数目 gi
此时我们在这样的条件下进行采样,缩小网络设计空间:
- di ≤ 16
- wi ≤ 1024 (其中 wi 可被 8 整除)
- bi ∈ {1, 2, 4}
- gi ∈ {1, 2, . . . , 32}
因此我们在 AnyNetX 衍生出其他搜索空间
- AnyNetXa 就是原始的 AnyNetX
- AnyNetXb 在 AnyNetX 基础上,每个 stage 使用相同的 Bottleneck 缩放比例 bi。并且实验得出缩放比例 bi <= 2 时最佳,参考下图最右边子图

- AnyNetXc 在 AnyNetXb 的基础上共享相同的分组数目 gi。由上图的左图和中间图可得知,从 A->C 的阶段,EDF 并没有受到影响,而我们此时已经减少了
- AnyNetXd 在 AnyNetXc 的基础上逐步增加 Block 的宽度 wi。此时网络性能有明显提升!
- AnyNetXe 在 AnyNetXd 的基础上在除了最后一个 stage 上,逐步增加 Block 的数量(深度)di。网络性能略微有提升
Swin Transformer v2
paper:https://arxiv.org/pdf/2111.09883.pdf
Swin Transformer V2: Scaling Up Capacity and Resolution:扩展容量和分辨率
Transformer 是 Google 的团队在 2017 年提出的一种 NLP 经典模型,现在比较火热的 Bert 也是基于 Transformer。Transformer 模型使用了 Self-Attention 机制,不采用 RNN 的顺序结构,使得模型可以并行化训练,而且能够拥有全局信息。
本文介绍这篇文章是 Swin Transformer 系列的升级版 Swin Transformer v2。Swin Transformer 是屠榜各大CV任务的通用视觉Transformer模型,它在图像分类、目标检测、分割上全面超越 SOTA,在语义分割任务中在 ADE20K 上刷到 53.5 mIoU,超过之前 SOTA 大概 4.5 mIoU!可能是CNN的完美替代方案。除此之外,本文一并介绍 Swin MLP 的代码实现,Swin Transformer 作者们在已有模型的基础上实现了 Swin MLP 模型,证明了 Window-based attention 对于 MLP 模型的有效性。
Swin Transformer Block 有两种,大致结构和 Transformer Block 一致,只是内部 attention 模块分别是 Window-based MSA 和 Shifted Window-based MSA。Window-based MSA 不同于普通的 MSA,它在一个个 window 里面去计算 self-attention,计算量与序列长度 N=hw 成线性关系。Window-based MSA 虽然大幅节约了计算量,但是牺牲了 windows 之间关系的建模,不重合的 Window 之间缺乏信息交流影响了模型的表征能力。Shifted Window-based MSA 就是为了解决这个问题。将下一层 Swin Transformer Block 的 Window 位置进行移动,得到不重合的 patch。
在 Swin Transformer 的基础上,研究人员进一步开发出了用于底层复原任务的 SwinIR
Swin Transformer v2 原理分析:
Swin Transformer 提出了一种针对视觉任务的通用的 Transformer 架构,MSRA 进一步打造了一个包含3 billion 个参数,且允许输入分辨率达到1560×1560的大型 Swin Transformer,称之为 SwinV2。它在多个基准数据集 (包含 ImageNet 分类、COCO 检测、ADE20K 语义分割以及Kinetics-400 动作分类) 上取得新记录,分别是 ImageNet 图像分类84.0% Top-1 accuracy,COCO 目标检测63.1/54.4 box / mask mAP,ADE20K 语义分割59.9mIoU,Kinetics-400视频动作识别86.8% Top-1 accuracy。
Swin Transformer v2 的核心目的是把 Swin Transformer 模型做大,做成类似 BERT large 那样包含 340M 参数的预训练大模型。在 NLP 中,有的预训练的大模型,比如 Megatron-Turing-530B 或者 Switch-Transformer-1.6T,参数量分别达到了530 billion 或者1.6 trillion。
另一方面,视觉大模型的发展却滞后了。 Vision Transformer 的大模型目前也只是达到了1-2 billion 的参数量,且只支持图像识别任务。部分原因是因为在训练和部署方面存在以下困难:
- 问题1:训练中的不稳定性问题。在大型模型中,跨层激活函数输出的幅值的差异变得更大。激活值是逐层累积的,因此深层的幅值明显大于浅层的幅值。如下图1所示是扩大模型容量时的不稳定问题。 当我们将原来的 Swin Transformer 模型从小模型放大到大模型时,深层的 activation 值急剧增加。最高和最低幅值之间的差异达到了104。当我们进一步扩展到一个巨大的规模 (658M 参数) 时,它不能完成训练,如图2所示。

- 问题2:许多下游视觉任务需要高分辨率的图像或窗口,预训练模型时是在低分辨率下进行的,而 fine-tuning 是在高分辨率下进行的。针对分辨率不同的问题传统的做法是把位置编码进行双线性插值 (bi-cubic interpolation),这种做法是次优的。如下图3所示是不同位置编码方式性能的比较,当我们直接在较大的图像分辨率和窗口大小测试预训练的 Imagenet-1k 模型 (分辨率256×256,window siez=8×8) 时,发现精度显着下降。

- 问题3:当图像分辨率较高时,GPU 内存消耗也是一个问题。
为了解决以上几点问题,作者提出了:
方法1:post normalization 技术:解决训练中的不稳定性问题。
把 Layer Normalization 层放在 Attention 或者 MLP 的后面。这样每个残差块的输出变化不至于太大,因为主分支和残差分支都是 LN 层的输出,有 LN 归一化作用的限制。如上图1所示,这种做法使得每一层的输出值基本上相差不大。在最大的模型训练中,作者每经过6个 Transformer Block,就在主支路上增加了一层 LN,以进一步稳定训练和输出幅值。
方法2:scaled cosine attention 技术:解决训练中的不稳定性问题。
原来的 self-attention 计算中,query 和 key 之间的相似性通过 dot-product 来衡量,作者发现这样学习到的 attention map 往往被少数像素对所支配。所以把 dot-product 改成了 cosine 函数,通过它来衡量 query 和 key 之间的相似性。
\[\operatorname{Sim}\left(\mathbf{q}i, \mathbf{k}_j\right)=\cos \left(\mathbf{q}_i, \mathbf{k}_j\right) / \tau+B{i j}\]
式中, \(B_{i j}\) 是下面讲得相对位置编码, \(\tau\) 是可学习参数。余弦函数是 naturally normalized,因 此可以有较温和的注意力值。
方法3:对数连续位置编码技术:解决分辨率变化导致的位置编码维度不一致问题。
- 该方法可以 更平滑地传递在低分辨率下预先训练好的模型权值,以处理高分辨率的模型权值。
我们首先复习下 Swin Transformer 的相对位置编码技术。
\[\operatorname{Attention}(Q, K, V)=\operatorname{SoftMax}\left(Q K^T / \sqrt{d}+B\right) V\]
式中, \(B \in \mathbb{R}^{M^2 \times M^2}\) 是每个 head 的相对位置偏差项 (relative position bias),\(Q, K, V \in \mathbb{R}^{M^2 \times d}\) 是 window-based attention 的 query, key 和 value。 window 的大小。
作者引入对数空间连续位置偏差 (log-spaced continuous position bias),使相对位置偏差在不同的 window 分辨率之下可以较为平滑地过渡。
方法4:节省 GPU memory 的方法:
1 Zero-Redundancy Optimizer (ZeRO) 技术:
来自论文:Zero: Memory optimizations toward training trillion parameter models
传统的数据并行训练方法 (如 DDP) 会把模型 broadcast 到每个 GPU 里面,这对于大型模型来讲非常不友好,比如参数量为 3,000M=3B 的大模型来讲,若使用 AdamW optimizer,32为的浮点数,就会占用 48G 的 GPU memory。通过使用 ZeRO optimizer, 将模型参数和相应的优化状态划分并分布到多个 GPU 中,从而大大降低了内存消耗。训练时使用 DeepSpeed framework,ZeRO stage-1 option。
2 Activation check-pointing 技术:
来自论文:Training deep nets with sublinear memory cost
Transformer 层中的特征映射也消耗了大量的 GPU 内存,在 image 和 window 分辨率较高的情况下会成为一个瓶颈。这个优化最多可以减少30%的训练速度。
3 Sequential self-attention computation 技术:
在非常大的分辨率下训练大模型时,如分辨率为1535×1536,window size=32×32时,在使用了上述两种优化策略之后,对于常规的 GPU (40GB 的内存)来说,仍然是无法承受的。作者发现在这种情况下,self-attention 模块构成了瓶颈。为了解决这个问题,作者实现了一个 sequential 的 self-attention 计算,而不是使用以前的批处理计算方法。这种优化在前两个阶段应用于各层,并且对整体的训练速度有一定的提升。
在这项工作中,作者还一方面适度放大 ImageNet-22k 数据集5倍,达到7000万张带有噪声标签的图像。 还采用了一种自监督学习的方法来更好地利用这些数据。通过结合这两种策略,作者训练了一个30亿参数的强大的 Swin Transformer 模型刷新了多个基准数据集的指标,并能够将输入分辨率提升至1536×1536 (Nvidia A100-40G GPUs)。此外,作者还分享了一些 SwinV2 的关键实现细节,这些细节导致了 GPU 内存消耗的显着节省,从而使得使用常规 GPU 来训练大型视觉模型成为可能。 作者的目标是在视觉预训练大模型这个方向上激发更多的研究,从而最终缩小视觉模型和语言模型之间的容量差距。
不同 Swin V2 的模型配置:
- SwinV2-T: C= 96, layer numbers ={2,2,6,2}
- SwinV2-S: C= 96, layer numbers ={2,2,18,2}
- SwinV2-B: C= 128, layer numbers ={2,2,18,2}
- SwinV2-L: C= 192, layer numbers ={2,2,18,2}
- SwinV2-H: C= 352, layer numbers ={2,2,18,2}
- SwinV2-G: C= 512, layer numbers ={2,2,42,2}
对于 SwinV2-H 和 SwinV2-G 的模型训练,作者每经过6个 Transformer Block,就在主支路上增加了一层 LN,以进一步稳定训练和输出幅值。
Experiments
模型:SwinV2-G,3B parameters
Image classification
Dataset for Evaluation:ImageNet-1k,ImageNet-1k V2
Dataset for Pre-Training:ImageNet-22K-ext (70M images, 22k classes)
训练策略:分辨率使用192×192,为了节约参数量。2-step 的预训练策略。首先以自监督学习的方式在 ImageNet-22K-ext 数据集上训练 20 epochs,再以有监督学习的方式在这个数据集上训练 30 epochs,SwinV2-G 模型在 ImageNet-1k 上面达到了惊人的90.17%的 Top-1 Accuracy,在 ImageNet-1k V2 上面也达到了惊人的84.00%的 Top-1 Accuracy,超过了历史最佳的83.33%。
同时,使用 Swin V2 的训练策略以后,Base 模型和 Large 模型的性能也可以进一步提升。比如 SwinV2-B 和 SwinV2-L 在 SwinV1-B 和 SwinV1-L 的基础上分别涨点0.8%和0.4%,原因来自更多的 labelled data (ImageNet-22k-ext, 70M images), 更强的 Regularization,或是自监督学习策略。
Object detection,Instance Segmentation
Dataset for Evaluation:COCO
Dataset for Pre-Training:Object 365 v2
如下图6所示 SwinV2-G 模型与之前在 COCO 目标检测和实例分割任务上取得最佳性能模型进行了比较。SwinV2-G 在 COCO test-dev 上实现了 63.1/54.4 box/max AP,相比于 SoftTeacher (61.3/53.0) 提高了 + 1.8/1.4。
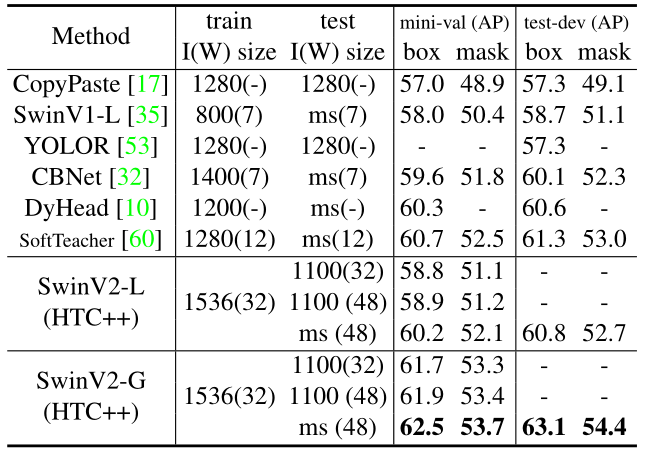
Semantic segmentation
Dataset for Evaluation:ADE20K
如下图7所示 SwinV2-G 模型与之前在 ADE20K 语义分割基准上的 SOTA 结果进行了比较。Swin-V2-G 在 ADE20K val 集上实现了 59.9 mIoU,相比于 BEiT 的 58.4 高了 1.5。
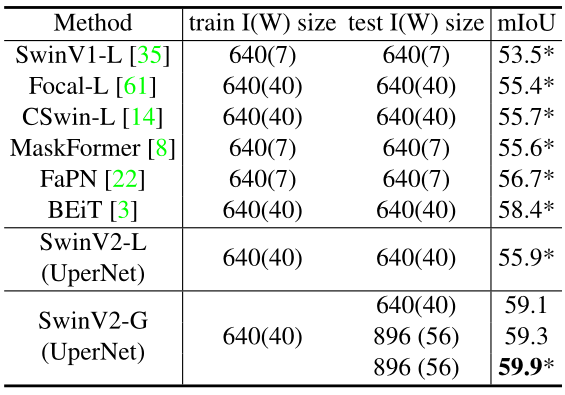
Video action classification
Dataset for Evaluation:Kinetics-400 (K400)
如下图8所示 SwinV2-G 模型与之前在 Kinetics-400 动作分类基准上的 SOTA 结果进行了比较。可以看到,Video-SwinV2-G 实现了 86.8% 的 top-1 准确率,比之前的 TokenLearner 方法的 85.4% 高出 +1.4%。
对比实验:post-norm 和 scaled cosine attention 的作用
如下图9所示,这两种技术均能提高 Swin-T,Swin-S 和 Swin-B 的性能,总体提高分别为 0.2%,0.4% 和 0.5%。说明该技术对大模型更有利。更重要的是,它们能让训练更稳定。对于 Swin-H 和 Swin-G 模型而言,自监督预训练使用原来的 Swin V1 无法收敛,而 Swin V2 模型训练得很好。